Introduction à Python
Charles Martin
Octobre 2022
Mise en place
Python est un language de programmation général. Contrairement à R ou à MATLAB, il n'a pas été conçu spécifiquement pour l'analyse et la visualisation de données. C'est pourquoi la plupart du code que nous utiliserons aujourd'hui provient de librairies de codes ayant été créées pour ces usages spécifiques.
Bien que beaucoup de systèmes d'exploitation sont livrés avec une version de Python pré-installée (entre autres Ubuntu, MacOS, etc.), sa configuration, l'installation de libraries, etc. peut être relativement périlleux pour un ou une débutante.
C'est pourquoi pour cet atelier, nous travaillerons plutôt avec Python sur Google Colab (https://colab.research.google.com/). Colab est un service offert par Google, où on peut se créer un cahier de notes (Jupyter Notebook) dans lequel on peut mélanger du code (entre autres en Python) et du texte (sous format Markdown). Comme tout les logiciels nécessaires sont déjà pré-installés, nous économiserons BEAUCOUP de temps de configuration.
On peut d'ailleurs, immédiatement, vérifier que notre cahier de notes fonctionne bien en lançant une première commande de test
print("Hello, World!")Hello, World!
Cela dit, si jamais vous envie de travailler directement sur vos ordinateurs, je vous conseille d'installer Python à travers un gestionnaire de librairies (par exemple Anaconda). Cette façon de faire vous permettra, entre autres, d'éviter les conflits de versions entre vos différents projets.
Les bases de Python
Python peut s'utiliser comme une calculatrice
1+123*515Les exposants sont différents de ceux que l'on voit souvent (i.e. 2^5)
2**8256On peut garder nos résultats dans un objet
mon_resultat = 3+2mon_resultat*630Les listes
Nous n'aurons ici (évidemment!) pas le temps de tout explorer les structures de données et fonctions possibles de Python.
Nous prendrons tout de même le temps d'en apprivoiser une, qui devrait être la plus utile, soit les listes
especes = ['bouleau','érable','chêne']On peut accéder aux divers éléments d'une liste à partir de leur index, qui commence à 0 (plutôt qu'à 1, c'est une vieille guerre de programmeurs)
print(especes[0])bouleau
On peut aussi modifier les éléments d'une liste
especes[2] = 'sapin'
print(especes)['bouleau', 'érable', 'sapin']
En ajouter, etc.
especes.append('épinette')
print(especes)['bouleau', 'érable', 'sapin', 'épinette']
Remarquez dans l'exemple précédent l'utilisation de la syntaxe object.fonction.
Nous n'irons pas trop creux dans ces détails aujourd'hui, mais puisque Python est un language orienté-objet, chacun des objets possède une série d'actions (des fonctions) qu'ils peuvent accomplir et de données associées. Pour accédér à ces derniers, on utilise la notation objet.méthode ou objet.donnée.
On peut aussi supprimer des items, soit par position, ou par leur valeur directement
del especes[1]
especes.remove('bouleau')
print(especes)['sapin', 'épinette']
Remarquez que si vous utilisez la méthode par valeur, seule la première valeur sera supprimée si vous travaillez de cette façon.
Les boucles et les fonctions
Pour automatiser les choses en Python, on peut entre autres, utiliser des fonctions et des boucles
On peut par exemple imprimer le nom de chacune des espèces comme ceci
for espece in especes :
print(espece)sapin
épinette
Remarquez que, dans Python, les espaces devant le code sont conventionnés et importants.
Tout le texte qui est en retrait fait partie de la boucle. Pas besoin d'accolades, de parenthèses, etc. C'est très élégant!
On peut aussi se créer une fonction qui, par exemple, vérifie si notre espèce est une épinette
def cest_une_epinette(x) :
if x == 'épinette' :
return "oui"
else :
return "non"for espece in especes :
print(espece)
print(cest_une_epinette(espece))sapin
non
épinette
oui
Les dictionnaires de données
Une autre structure de données importante de Python se nomme les dictionnaires de données. Alors que les listes ne contiennent que des séries de valeurs, les dictionnaires contiennent des paires d'associations clé-valeur.
Ils sont définis en utilisant les accolades plutôt que les parenthèses carrées.
infos = {'nom' : 'charles', 'age' : 42, 'ville': 'Trois-Rivières'}
infos{'nom': 'charles', 'age': 42, 'ville': 'Trois-Rivières'}On accède tout de même aux éléments d'un dictionnaire à l'aide des parenthèses carrées
infos['nom']{"type":"string"}On peut obtenir l'ensemble des clés ou des valeurs comme ceci :
print(infos.keys())
print(infos.values())dict_keys(['nom', 'age', 'ville'])
dict_values(['charles', 42, 'Trois-Rivières'])
Il est possible de combiner différentes structures de données pour former des objets complexes, comme par exemple une liste de dictionnaires :
repondants = [
{'nom' : 'Charles', 'language' : 'R'},
{'nom' : 'Alex', 'language' : 'Python'}
]
for repondant in repondants :
print(repondant['nom']+' préfère ' + repondant['language'])Charles préfère R
Alex préfère Python
Vectorisation des opérations
Contrairement à plusieurs languages créés expressément pour l'analyse de données, Python n'est pas un language naturellement vectorisé.
Si on se prépare une liste et que l'on veut multiplier tous ses items par 2, on ne peut PAS faire ceci :
liste = [1,2,3,4]liste*2[1, 2, 3, 4, 1, 2, 3, 4]On pourrait évidemment contourner le problème avec des boucles
nouvelle_liste = []
for chiffre in liste :
nouvelle_liste.append(chiffre*2)
nouvelle_liste[2, 4, 6, 8]Python possède une notation plus compacte pour ce genre de problèmes, que l'on pourrait traduire par les listes en compréhension (List Comprehension)
[x * 2 for x in liste][2, 4, 6, 8]Il s'agit d'une structure très compacte, permettant de créer une liste à partir d'une autre liste.
Enfin, si vous avez beaucoup d'algèbre vectoriel ou matriciel à faire, il sera probablement plus simple d'utiliser la librairie prévue à cet effet, qui se nomme NumPy.
NumPy (et toutes les autres librairies nécessaires à l'atelier) sont déjà installées sur Colab.
Sur vos ordinateurs personnels, on installe NumPy par la ligne de commande (terminal), par exemple comme ceci :
pip install numpy
Dans Python, lorsque l'on active une librarie, tout le code chargé est associé à un préfixe, que l'on peut modifier au moment de l'activation
import numpy as np
tableau = np.array([1,2,3,4])
print(tableau)
print(tableau*2)[1 2 3 4]
[2 4 6 8]
matrice = np.array([[1,2,3,4],[5,6,7,8]])
print(matrice)[[1 2 3 4]
[5 6 7 8]]
print(matrice.dot(tableau))[30 70]
Les tableaux de données pandas
Il existe une librairie gérant les tableaux de données de façon beaucoup plus appropriée que les listes ou les dictionnaires lorsqu'il s'agit de l'analyse statistiques de données. Cette librairie se nomme pandas.
Elle tient son nom d'une structure de données commune en économie, les PANel DAta.
import pandas as pdCharger et vérifier les données
La fonction de pandas pour charger des données se nomme read_csv.
Pour se simplifier la vie, nous allons charger des données directement disponibles sur le web
iris = pd.read_csv('https://raw.githubusercontent.com/mwaskom/seaborn-data/master/iris.csv')
iris| sepal_length | sepal_width | petal_length | petal_width | species | |
|---|---|---|---|---|---|
| 0 | 5.1 | 3.5 | 1.4 | 0.2 | setosa |
| 1 | 4.9 | 3.0 | 1.4 | 0.2 | setosa |
| 2 | 4.7 | 3.2 | 1.3 | 0.2 | setosa |
| 3 | 4.6 | 3.1 | 1.5 | 0.2 | setosa |
| 4 | 5.0 | 3.6 | 1.4 | 0.2 | setosa |
| ... | ... | ... | ... | ... | ... |
| 145 | 6.7 | 3.0 | 5.2 | 2.3 | virginica |
| 146 | 6.3 | 2.5 | 5.0 | 1.9 | virginica |
| 147 | 6.5 | 3.0 | 5.2 | 2.0 | virginica |
| 148 | 6.2 | 3.4 | 5.4 | 2.3 | virginica |
| 149 | 5.9 | 3.0 | 5.1 | 1.8 | virginica |
150 rows × 5 columns
Si jamais vos données sont en format Excel, il existe aussi une fonction read_excel permettant de les lire directement, sans passer par le format CSV.
Une fois les données chargées, il est important de vérifier le contenu de notre tableau de données, par exemple avec la méthode info :
iris.info()<class 'pandas.core.frame.DataFrame'>
RangeIndex: 150 entries, 0 to 149
Data columns (total 5 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 sepal_length 150 non-null float64
1 sepal_width 150 non-null float64
2 petal_length 150 non-null float64
3 petal_width 150 non-null float64
4 species 150 non-null object
dtypes: float64(4), object(1)
memory usage: 6.0+ KB
Trier les observations
En ordre croissant
iris.sort_values('sepal_length')| sepal_length | sepal_width | petal_length | petal_width | species | |
|---|---|---|---|---|---|
| 13 | 4.3 | 3.0 | 1.1 | 0.1 | setosa |
| 42 | 4.4 | 3.2 | 1.3 | 0.2 | setosa |
| 38 | 4.4 | 3.0 | 1.3 | 0.2 | setosa |
| 8 | 4.4 | 2.9 | 1.4 | 0.2 | setosa |
| 41 | 4.5 | 2.3 | 1.3 | 0.3 | setosa |
| ... | ... | ... | ... | ... | ... |
| 122 | 7.7 | 2.8 | 6.7 | 2.0 | virginica |
| 118 | 7.7 | 2.6 | 6.9 | 2.3 | virginica |
| 117 | 7.7 | 3.8 | 6.7 | 2.2 | virginica |
| 135 | 7.7 | 3.0 | 6.1 | 2.3 | virginica |
| 131 | 7.9 | 3.8 | 6.4 | 2.0 | virginica |
150 rows × 5 columns
Ou en ordre décroissant
iris.sort_values('sepal_length', ascending = False)| sepal_length | sepal_width | petal_length | petal_width | species | |
|---|---|---|---|---|---|
| 131 | 7.9 | 3.8 | 6.4 | 2.0 | virginica |
| 135 | 7.7 | 3.0 | 6.1 | 2.3 | virginica |
| 122 | 7.7 | 2.8 | 6.7 | 2.0 | virginica |
| 117 | 7.7 | 3.8 | 6.7 | 2.2 | virginica |
| 118 | 7.7 | 2.6 | 6.9 | 2.3 | virginica |
| ... | ... | ... | ... | ... | ... |
| 41 | 4.5 | 2.3 | 1.3 | 0.3 | setosa |
| 42 | 4.4 | 3.2 | 1.3 | 0.2 | setosa |
| 38 | 4.4 | 3.0 | 1.3 | 0.2 | setosa |
| 8 | 4.4 | 2.9 | 1.4 | 0.2 | setosa |
| 13 | 4.3 | 3.0 | 1.1 | 0.1 | setosa |
150 rows × 5 columns
##Sélectionner des lignes
Il existe plusieurs façon de sélectionner des lignes ou des colonnes avec pandas, mais la façon la plus générale est d'utiliser l'opérateur loc.
Ce dernier peut accepter soit un liste de valeurs booléennes, soit des étiquettes ou une séquence entre deux étiquettes.
Cet opérateur vous permet aussi de sélectionner simultanément une liste de colonnes ET de lignes dans la même opération.
iris.loc[iris.species == "virginica"]| sepal_length | sepal_width | petal_length | petal_width | species | |
|---|---|---|---|---|---|
| 100 | 6.3 | 3.3 | 6.0 | 2.5 | virginica |
| 101 | 5.8 | 2.7 | 5.1 | 1.9 | virginica |
| 102 | 7.1 | 3.0 | 5.9 | 2.1 | virginica |
| 103 | 6.3 | 2.9 | 5.6 | 1.8 | virginica |
| 104 | 6.5 | 3.0 | 5.8 | 2.2 | virginica |
| 105 | 7.6 | 3.0 | 6.6 | 2.1 | virginica |
| 106 | 4.9 | 2.5 | 4.5 | 1.7 | virginica |
| 107 | 7.3 | 2.9 | 6.3 | 1.8 | virginica |
| 108 | 6.7 | 2.5 | 5.8 | 1.8 | virginica |
| 109 | 7.2 | 3.6 | 6.1 | 2.5 | virginica |
| 110 | 6.5 | 3.2 | 5.1 | 2.0 | virginica |
| 111 | 6.4 | 2.7 | 5.3 | 1.9 | virginica |
| 112 | 6.8 | 3.0 | 5.5 | 2.1 | virginica |
| 113 | 5.7 | 2.5 | 5.0 | 2.0 | virginica |
| 114 | 5.8 | 2.8 | 5.1 | 2.4 | virginica |
| 115 | 6.4 | 3.2 | 5.3 | 2.3 | virginica |
| 116 | 6.5 | 3.0 | 5.5 | 1.8 | virginica |
| 117 | 7.7 | 3.8 | 6.7 | 2.2 | virginica |
| 118 | 7.7 | 2.6 | 6.9 | 2.3 | virginica |
| 119 | 6.0 | 2.2 | 5.0 | 1.5 | virginica |
| 120 | 6.9 | 3.2 | 5.7 | 2.3 | virginica |
| 121 | 5.6 | 2.8 | 4.9 | 2.0 | virginica |
| 122 | 7.7 | 2.8 | 6.7 | 2.0 | virginica |
| 123 | 6.3 | 2.7 | 4.9 | 1.8 | virginica |
| 124 | 6.7 | 3.3 | 5.7 | 2.1 | virginica |
| 125 | 7.2 | 3.2 | 6.0 | 1.8 | virginica |
| 126 | 6.2 | 2.8 | 4.8 | 1.8 | virginica |
| 127 | 6.1 | 3.0 | 4.9 | 1.8 | virginica |
| 128 | 6.4 | 2.8 | 5.6 | 2.1 | virginica |
| 129 | 7.2 | 3.0 | 5.8 | 1.6 | virginica |
| 130 | 7.4 | 2.8 | 6.1 | 1.9 | virginica |
| 131 | 7.9 | 3.8 | 6.4 | 2.0 | virginica |
| 132 | 6.4 | 2.8 | 5.6 | 2.2 | virginica |
| 133 | 6.3 | 2.8 | 5.1 | 1.5 | virginica |
| 134 | 6.1 | 2.6 | 5.6 | 1.4 | virginica |
| 135 | 7.7 | 3.0 | 6.1 | 2.3 | virginica |
| 136 | 6.3 | 3.4 | 5.6 | 2.4 | virginica |
| 137 | 6.4 | 3.1 | 5.5 | 1.8 | virginica |
| 138 | 6.0 | 3.0 | 4.8 | 1.8 | virginica |
| 139 | 6.9 | 3.1 | 5.4 | 2.1 | virginica |
| 140 | 6.7 | 3.1 | 5.6 | 2.4 | virginica |
| 141 | 6.9 | 3.1 | 5.1 | 2.3 | virginica |
| 142 | 5.8 | 2.7 | 5.1 | 1.9 | virginica |
| 143 | 6.8 | 3.2 | 5.9 | 2.3 | virginica |
| 144 | 6.7 | 3.3 | 5.7 | 2.5 | virginica |
| 145 | 6.7 | 3.0 | 5.2 | 2.3 | virginica |
| 146 | 6.3 | 2.5 | 5.0 | 1.9 | virginica |
| 147 | 6.5 | 3.0 | 5.2 | 2.0 | virginica |
| 148 | 6.2 | 3.4 | 5.4 | 2.3 | virginica |
| 149 | 5.9 | 3.0 | 5.1 | 1.8 | virginica |
Remarquez que, si on accède à une seule colonne à la fois et que le nom de la colonne est un identifiant valide de Python (pas d'espace, pas d'accents, etc.), on peut l'accéder directement avec un .variable plutôt que ['variable']
Sélectionner des colonnes
Le principe est le même pour sélectionner des colonnes. On doit par contre à ce moment spécifier quelles lignes on veut obtenir (ou spécifier toutes les lignes, comme ceci)
iris.loc[:,["sepal_width", "species"]]| sepal_width | species | |
|---|---|---|
| 0 | 3.5 | setosa |
| 1 | 3.0 | setosa |
| 2 | 3.2 | setosa |
| 3 | 3.1 | setosa |
| 4 | 3.6 | setosa |
| ... | ... | ... |
| 145 | 3.0 | virginica |
| 146 | 2.5 | virginica |
| 147 | 3.0 | virginica |
| 148 | 3.4 | virginica |
| 149 | 3.0 | virginica |
150 rows × 2 columns
Et comme discuté, on peut combiner les deux opérations dans une même commande :
iris.loc[iris.species == "virginica",["sepal_width", "species"]]| sepal_width | species | |
|---|---|---|
| 100 | 3.3 | virginica |
| 101 | 2.7 | virginica |
| 102 | 3.0 | virginica |
| 103 | 2.9 | virginica |
| 104 | 3.0 | virginica |
| 105 | 3.0 | virginica |
| 106 | 2.5 | virginica |
| 107 | 2.9 | virginica |
| 108 | 2.5 | virginica |
| 109 | 3.6 | virginica |
| 110 | 3.2 | virginica |
| 111 | 2.7 | virginica |
| 112 | 3.0 | virginica |
| 113 | 2.5 | virginica |
| 114 | 2.8 | virginica |
| 115 | 3.2 | virginica |
| 116 | 3.0 | virginica |
| 117 | 3.8 | virginica |
| 118 | 2.6 | virginica |
| 119 | 2.2 | virginica |
| 120 | 3.2 | virginica |
| 121 | 2.8 | virginica |
| 122 | 2.8 | virginica |
| 123 | 2.7 | virginica |
| 124 | 3.3 | virginica |
| 125 | 3.2 | virginica |
| 126 | 2.8 | virginica |
| 127 | 3.0 | virginica |
| 128 | 2.8 | virginica |
| 129 | 3.0 | virginica |
| 130 | 2.8 | virginica |
| 131 | 3.8 | virginica |
| 132 | 2.8 | virginica |
| 133 | 2.8 | virginica |
| 134 | 2.6 | virginica |
| 135 | 3.0 | virginica |
| 136 | 3.4 | virginica |
| 137 | 3.1 | virginica |
| 138 | 3.0 | virginica |
| 139 | 3.1 | virginica |
| 140 | 3.1 | virginica |
| 141 | 3.1 | virginica |
| 142 | 2.7 | virginica |
| 143 | 3.2 | virginica |
| 144 | 3.3 | virginica |
| 145 | 3.0 | virginica |
| 146 | 2.5 | virginica |
| 147 | 3.0 | virginica |
| 148 | 3.4 | virginica |
| 149 | 3.0 | virginica |
Remarquez que le tableau original n'est pas affecté par ces opérations. Si on veut conserver le résultat, on doit le garder dans un objet
iris.shape(150, 5)iris_modifie = iris.loc[iris.species == "virginica",["sepal_width", "species"]]iris.shape(150, 5)iris_modifie.shape(50, 2)##Ajouter des colonnes
iris.assign(ratio = iris.sepal_length / iris.petal_length)| sepal_length | sepal_width | petal_length | petal_width | species | ratio | |
|---|---|---|---|---|---|---|
| 0 | 5.1 | 3.5 | 1.4 | 0.2 | setosa | 3.642857 |
| 1 | 4.9 | 3.0 | 1.4 | 0.2 | setosa | 3.500000 |
| 2 | 4.7 | 3.2 | 1.3 | 0.2 | setosa | 3.615385 |
| 3 | 4.6 | 3.1 | 1.5 | 0.2 | setosa | 3.066667 |
| 4 | 5.0 | 3.6 | 1.4 | 0.2 | setosa | 3.571429 |
| ... | ... | ... | ... | ... | ... | ... |
| 145 | 6.7 | 3.0 | 5.2 | 2.3 | virginica | 1.288462 |
| 146 | 6.3 | 2.5 | 5.0 | 1.9 | virginica | 1.260000 |
| 147 | 6.5 | 3.0 | 5.2 | 2.0 | virginica | 1.250000 |
| 148 | 6.2 | 3.4 | 5.4 | 2.3 | virginica | 1.148148 |
| 149 | 5.9 | 3.0 | 5.1 | 1.8 | virginica | 1.156863 |
150 rows × 6 columns
Résumer les données
Pour avoir un aperçu de l'ensemble des statistiques descriptives de notre tableau de données, on peut utiliser la fonction describe
iris.describe()| sepal_length | sepal_width | petal_length | petal_width | |
|---|---|---|---|---|
| count | 150.000000 | 150.000000 | 150.000000 | 150.000000 |
| mean | 5.843333 | 3.057333 | 3.758000 | 1.199333 |
| std | 0.828066 | 0.435866 | 1.765298 | 0.762238 |
| min | 4.300000 | 2.000000 | 1.000000 | 0.100000 |
| 25% | 5.100000 | 2.800000 | 1.600000 | 0.300000 |
| 50% | 5.800000 | 3.000000 | 4.350000 | 1.300000 |
| 75% | 6.400000 | 3.300000 | 5.100000 | 1.800000 |
| max | 7.900000 | 4.400000 | 6.900000 | 2.500000 |
Si on a besoin de résumer nos données de façon plus fine, on peut spécifier les fonctions et les colonnes manuellement
iris.loc[:,['sepal_length', 'petal_length']].agg(['mean','min','max','count'])| sepal_length | petal_length | |
|---|---|---|
| mean | 5.843333 | 3.758 |
| min | 4.300000 | 1.000 |
| max | 7.900000 | 6.900 |
| count | 150.000000 | 150.000 |
On peut aussi appliquer nos résumés sur des groupes en particulier
iris.groupby('species')[['sepal_length', 'petal_length']].mean()| sepal_length | petal_length | |
|---|---|---|
| species | ||
| setosa | 5.006 | 1.462 |
| versicolor | 5.936 | 4.260 |
| virginica | 6.588 | 5.552 |
Visualisation avec matplotlib
Le module classique pour la visualisation de données avec Python se nomme matplotlib. En général, les gens utilisent en particulier la collection de fonctions nommée pyplot, qui vise à reproduire l'interface graphique de MATLAB dans Python
import matplotlib.pyplot as pltExplorer chaque variable individuellement
Pour créer un histogramme de fréquences, la fonction se nomme hist.
plt.hist('sepal_length', data = iris)(array([ 9., 23., 14., 27., 16., 26., 18., 6., 5., 6.]),
array([4.3 , 4.66, 5.02, 5.38, 5.74, 6.1 , 6.46, 6.82, 7.18, 7.54, 7.9 ]),
<a list of 10 Patch objects>)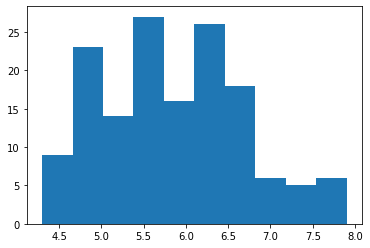
Comme matplotlib et pandas sont extrêmement bien intégrés, il existe, pour la plupart des visualisations, une façon de les appeller directement à partir du tableau de données
iris.sepal_length.plot.hist()<matplotlib.axes._subplots.AxesSubplot at 0x7f17e8c77a50>
On peut ensuite apporter des modifications à ce graphique (ou au précédent), puis demander à le réafficher
iris.sepal_length.plot.hist()
plt.ylabel("Fréquence")
plt.xlabel("Longueur des sépales")Text(0.5, 0, 'Longueur des sépales')
Si jamais vous travaillez avec matplotlib dans un script plutôt que sur Jupyter, vous devrez appeller plt.show() pour afficher votre graphique. Ici, Jupyter le fait pour vous.
Si nos données sont qualitatives, on emploie plutôt un diagramme à bandes.
Il faut par contre calculer d'abord nous même les dénombremenets de chacune des classes
iris.species.value_counts().plot.bar()<matplotlib.axes._subplots.AxesSubplot at 0x7f17e8b20150>
Explorer les relations entre deux variables
Pour regarder la relation entre deux variables quantiatives, on a bien sûr accès au classique nuage de points
iris.plot.scatter('petal_length', 'petal_width')<matplotlib.axes._subplots.AxesSubplot at 0x7f17e8af49d0>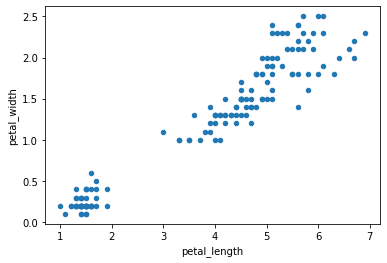
Et je vous montre rapidement comment changer la couleur des points parce que j'ai eu un peu de difficulté à trouver comment pour une variable qualitative.
La clé est de d'abord se créer un dictionnaire associant chacune des espèces à une couleur.
Puis au moment de créer le graphique, d'associer chaque valeur d'espèce à une valeur de couleur avec la méthode map.
couleurs = {'setosa':'red', 'virginica':'green', 'versicolor':'blue'}
iris.plot.scatter('petal_length','petal_width',c=iris.species.map(couleurs))<matplotlib.axes._subplots.AxesSubplot at 0x7f17e8a8be90>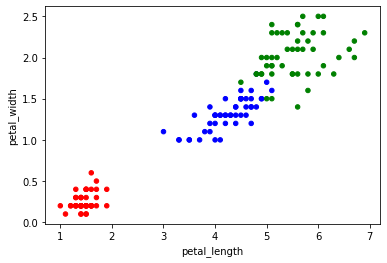
Si on veut explorer la relation entre une variable qualitative et une quantitative, il y a évidemment le boxplot
iris.boxplot(column='sepal_length',by='species', grid = False)<matplotlib.axes._subplots.AxesSubplot at 0x7f17e89f8150>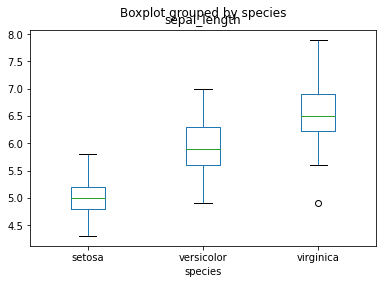
Un petit clin d'oeil à R
Si jamais vous vous ennuyez de ggplot2, l'ensemble de la librairie a été ré-implémentée en Python dans la librairie plotnine.
Remarquez qu'il existe tout de même certaines petites nuances vs. le code R. Vous ne pourrez pas copier-coller directement votre code.
from plotnine import ggplot, aes,geom_point
ggplot(iris) + aes(x = "sepal_length", y = "sepal_width") + geom_point()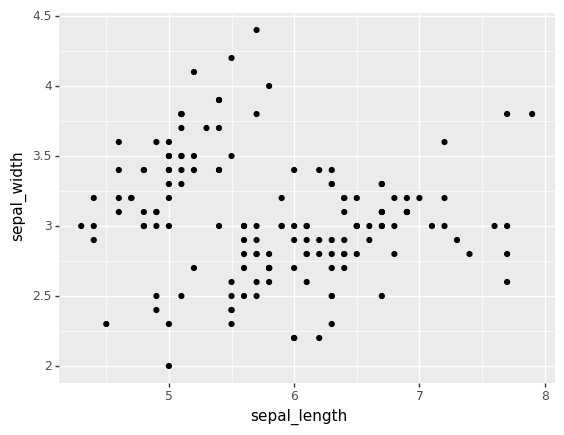
<ggplot: (8733791616949)>Tests statistiques avec scipy
Les tests statistiques classiques dans Python sont fournis par la collection de fonctions stats dans le module scipy
from scipy import statsSi vous voulez par exemple appliquer un test de T pour variance inégales
stats.ttest_ind(
iris.loc[iris.species == "virginica", 'petal_length'],
iris.loc[iris.species == "setosa", 'petal_length'],
equal_var = False
)Ttest_indResult(statistic=49.98618625709594, pvalue=9.26962758534569e-50)Si vous voulez mesurer une corrélationn de Pearson (et obtenir la valeur de p correspondante)
stats.pearsonr(
iris.petal_length,
iris.sepal_length
)(0.8717537758865831, 1.0386674194498099e-47)L'apprentissage automatique (Machine Learning) avec scikit-learn
La référence en matière d'aprentissage automatique (Machine Learning) est sans aucun doute le module Python nommé scikit-learn
Exemple avec un Random Forest
Voici comment on pourrait par exemple appliquer un modèle de classification Random Forest à nos données.
Remarquez qu'il faut d'abord encoder notre variable qualitative en numérique pour que le modèle la comprenne...
from sklearn.preprocessing import LabelEncoder
le = LabelEncoder()
Y = le.fit_transform(iris.species)
X = iris.loc[:,['sepal_length','sepal_width']]On peut ensuite lancer le modèle et voir à quel point cela fonctionne bien!
from sklearn.ensemble import RandomForestClassifier
rf = RandomForestClassifier()
rf.fit(X,Y)
rf.score(X,Y)0.9266666666666666Exemple avec une ACP
La librairie sklearn contient une fonction pour calculer une analyse en composantes principales nommé PCA dans le module decomposition. L'ACP se trouve à cet endroit, puisqu'il s'agit, dans le language de la science des données, d'une technique d'apprentissage non-supervisée.
Remarquez que la fonction applique toujours son calcul à la matrice de variance/covariance. Vous devrez vous-même centrer-réduire vos données pour utiliser la matrice de corrélation.
Une fonction pour effectuer cette opération (StandardScaler) se trouve dans le module preprocessing de sklearn.
from sklearn.decomposition import PCA
donnees_pour_acp = iris.drop(columns='species')
acp = PCA()
coordonnees_acp = acp.fit_transform(donnees_pour_acp)Voici comment récupérer les eigenvectors
acp.components_array([[ 0.36138659, -0.08452251, 0.85667061, 0.3582892 ],
[ 0.65658877, 0.73016143, -0.17337266, -0.07548102],
[-0.58202985, 0.59791083, 0.07623608, 0.54583143],
[-0.31548719, 0.3197231 , 0.47983899, -0.75365743]])Les eigenvalues
acp.explained_variance_array([4.22824171, 0.24267075, 0.0782095 , 0.02383509])Et la fraction de la variance expliquée par chacun des axes
acp.explained_variance_ratio_array([0.92461872, 0.05306648, 0.01710261, 0.00521218])Enfin, on peut aussi utiliser matplotlib pour visualiser les deux premiers axes de notre ACP comme ceci :
plt.scatter(coordonnees_acp[:,0], coordonnees_acp[:,1])<matplotlib.collections.PathCollection at 0x7f17d0756750>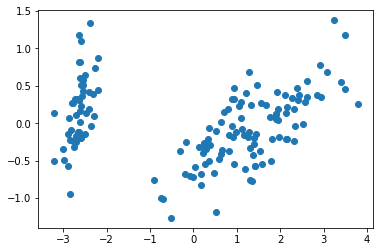
On peut aussi tracer les variables dans le nouveau système d'axes. Par contre, il faut utiliser une boucle pour y arriver, parce que la fonction plt.arrow ne trace qu'une flèche à la fois
PC1 = acp.components_[0]
PC2 = acp.components_[1]
for i in range(len(PC1)):
plt.arrow(0, 0, PC1[i], PC2[i], head_width = 0.05)
plt.text(PC1[i], PC2[i],list(donnees_pour_acp)[i])
plt.axvline(0, linestyle = "--")
plt.axhline(0, linestyle = "--")
plt.xlim(-1.5,1.5)
plt.ylim(-1,1)(-1.0, 1.0)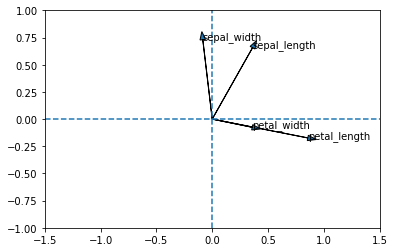
Comme ceci n'est pas un atelier de statistiques, je vous laisse le plaisir de mettre à l'échelle les deux données (eigenvectors et points) dans un même graphique avec le scaling de votre choix ;-)
Ressources
Voici en terminant deux ressources qui m'ont été utiles pour construire cet atelier :
Si vous voulez vraiment apprendre Python, le livre le plus recommandé à l'heure actuelle est Python Crash Course, 2nd edition, par Eric Matthes. Au delà des 200 premières pages vous enseignant les bases de Python, les 300 autres sont consacrées à 3 projets vous montrant les différentes facettes du language : un jeu comprenant une interface visuelle, un projet d'analyse de données et une application Web.
Si jamais vous voulez appliquer ce que vous avez vu dans vos cours de statistiques avec Python (et pour chaque bout de code voir l'équivalent en R), je vous conseille Practical Statistics for Data Scientists, Second Edition, par Bruce, Bruce et Gedeck. Il s'agit à la fois d'un des rares livres présentant ses exemples en deux languages et il offre, à mon avis, juste le bon niveau de détail pour quelqu'un qui veut appliquer correctement les bonnes techniques statistiques, sans avoir l'ambition de devenir statistien pour autant.