Visualisation rapide des données avec ggplot2
Charles Martin
Septembre 2018
- Clarification
- Conditions pour apprécier votre temps avec ggplot2
- Notre jeu de données
- Un premier graphique
- Quelles autres propriétés graphiques sont disponibles?
- Exemple
- Pour modifier l’ensemble des points
- Exercice #1
- Là où ggplot est particulièrement efficace
- Quelles couches graphiques sont disponibles?
- Si vous avez des millions de points
- Visualisation de l’incertitude
- Exercice #2
- L’outil d’exploration ultime
- Transformations rapides (sans toucher aux données)
- Avant de fignoler, apprendre à bien sauvegarder…
- Et finalement, le fameux fond gris…
Clarification
Tout ce que nous ferons aujourd’hui pourrait aussi s’accomplir avec les graphiques de base de R.
L’avantage de ggplot2 est qu’on passe moins de temps à programmer (création de boucles, gestion de tableaux, conditions etc.) et plus de temps à visualiser les données.
Conditions pour apprécier votre temps avec ggplot2
- Besoin de données rectangulaires et propres (tidy data)
- Ne combattez pas le paradigme, ggplot2 n’est pas la même chose que les graphiques de base avec d’autres noms
- Prenez le temps de bien comprendre la structure type d’un graphique ggplot2
- Imprimez l’aide-mémoire (cheat-sheet)
- N’ayez pas peur d’insérer des sauts de lignes et des tabulations pour clarifier votre code
Notre jeu de données
library(ggplot2)
Warning: package 'ggplot2' was built under R version 3.5.2
data(msleep)
summary(msleep)
name genus vore
Length:83 Length:83 Length:83
Class :character Class :character Class :character
Mode :character Mode :character Mode :character
order conservation sleep_total sleep_rem
Length:83 Length:83 Min. : 1.90 Min. :0.100
Class :character Class :character 1st Qu.: 7.85 1st Qu.:0.900
Mode :character Mode :character Median :10.10 Median :1.500
Mean :10.43 Mean :1.875
3rd Qu.:13.75 3rd Qu.:2.400
Max. :19.90 Max. :6.600
NA's :22
sleep_cycle awake brainwt bodywt
Min. :0.1167 Min. : 4.10 Min. :0.00014 Min. : 0.005
1st Qu.:0.1833 1st Qu.:10.25 1st Qu.:0.00290 1st Qu.: 0.174
Median :0.3333 Median :13.90 Median :0.01240 Median : 1.670
Mean :0.4396 Mean :13.57 Mean :0.28158 Mean : 166.136
3rd Qu.:0.5792 3rd Qu.:16.15 3rd Qu.:0.12550 3rd Qu.: 41.750
Max. :1.5000 Max. :22.10 Max. :5.71200 Max. :6654.000
NA's :51 NA's :27
83 observations tirées de la littérature sur le temps de sommeil des mammifères.
- Entre autres des mesures sur le sommeil (en heures) :
sleep_totalsleep_rem(temps de sommeil paradoxal)sleep_cycleawake
- Et sur d’autres caractéristiques (en kg) :
brainwtbodywt
Un premier graphique
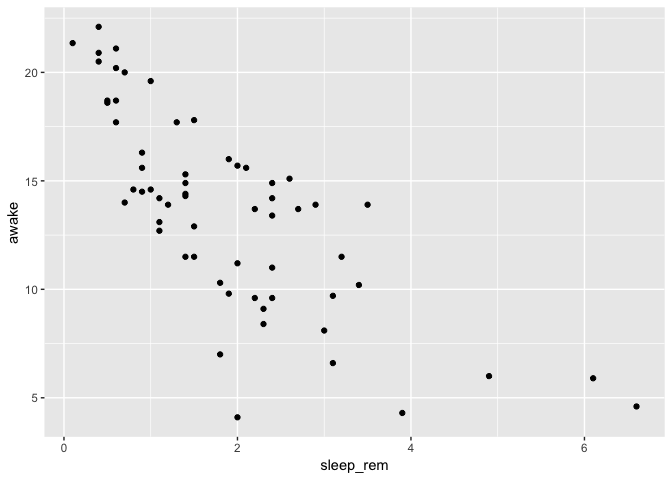
ggplot(data = msleep) +
geom_point(mapping = aes(x = sleep_rem, y = awake))
Warning: Removed 22 rows containing missing values (geom_point).
NB on peut insérer des sauts de lignes et des tabulations n’importe où dans le code R, du moment que c’est clair pour R qu’il faut qu’il attende la suite de l’instruction.
ggplot : créé l’objet graphique et y associe le tableau de données.
On ajoute ensuite des couches à cet objet.
mapping : crée l’association entre les variables du tableau de données
et les propriétés visuelles (aes) du graphique
Quelles autres propriétés graphiques sont disponibles?
Entre autres…
colorsizealpha(transparence, 0-1)shape(max 6)
L’association entre les propriétés (color et shape) et les valeurs dans le
tableau de données se nomme le “mapping”
Elles permettent d’ajouter des dimensions d’information supplémentaires, au delà de la position.
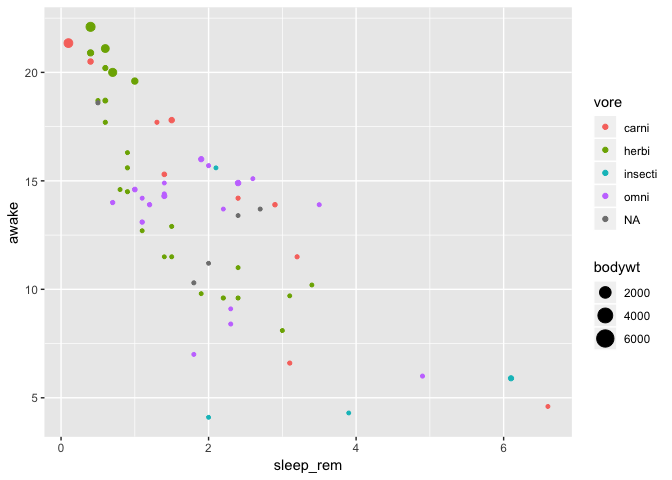
Exemple
Nous pouvons refaire le graphique précédent, mais en ajouter une couleur
par type d’alimentation (vore) et que la taille des points soit
proportionnelle à la
taille du corps de l’animal (bodywt)
ggplot(data = msleep) +
geom_point(mapping = aes(
x = sleep_rem,
y = awake,
color = vore,
size = bodywt
)
)
Warning: Removed 22 rows containing missing values (geom_point).
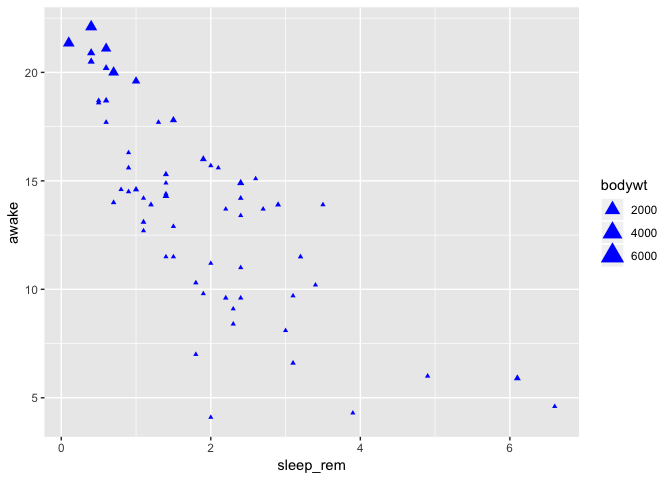
Pour modifier l’ensemble des points
Il faut spécifier la propriété visuelle à l’extérieur du bloc aes, p. ex.
ggplot(data = msleep) +
geom_point(
mapping = aes(
x = sleep_rem,
y = awake,
size = bodywt
),
color = "blue",
shape = 17
)
Warning: Removed 22 rows containing missing values (geom_point).
Exercice #1
Faites un graphique du poids du cerveau (brainwt) en fonction du poids total
de l’animal (bodywt)
Remplacez les points par des carrés
La couleur du point doit représenter le statut de conservation (conservation)
Là où ggplot est particulièrement efficace
Traçons d’abord un grahpique du temps d’éveil (awake) en fonction du
temps de sommeil paradoxal (sleep_rem).
ggplot(data = msleep) +
geom_point(mapping = aes(
x = sleep_rem,
y = awake
)
)
Warning: Removed 22 rows containing missing values (geom_point).
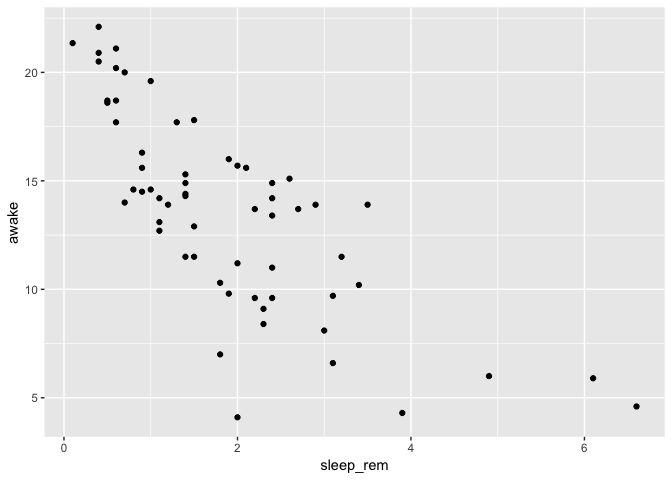
Ajouter une couleur par type d’alimentation (vore)
ggplot(data = msleep) +
geom_point(mapping = aes(
x = sleep_rem,
y = awake,
color = vore
)
)
Warning: Removed 22 rows containing missing values (geom_point).
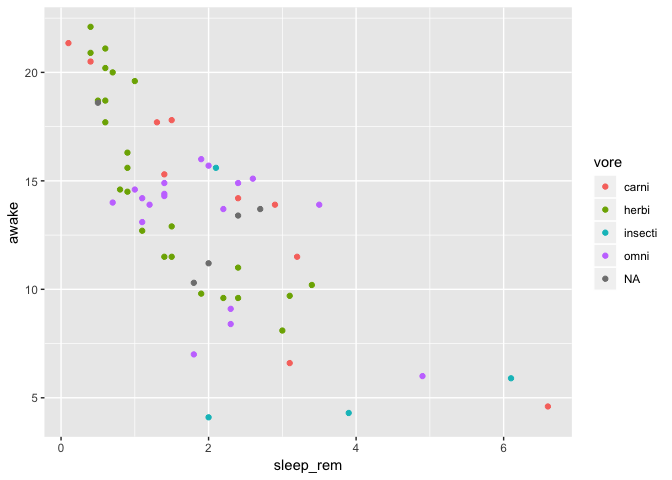
Maintenant un collègue passe derrière-vous et se demande si le graphique serait plus clair avec un panneau par type d’alimentation
ggplot(data = msleep) +
geom_point(mapping = aes(
x = sleep_rem,
y = awake
)) +
facet_wrap(~vore)
Warning: Removed 22 rows containing missing values (geom_point).
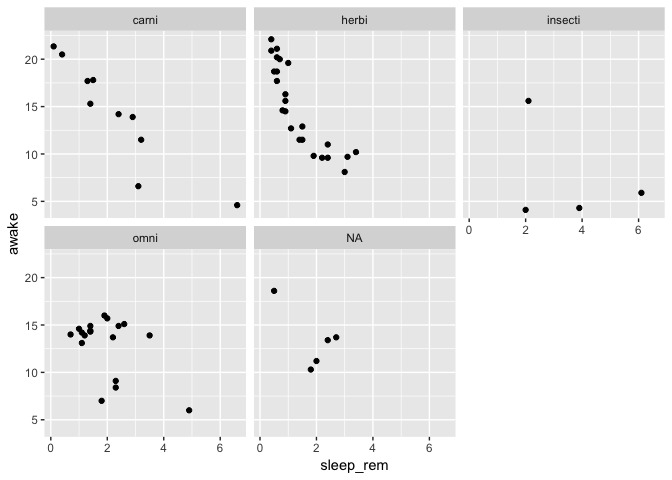
Ok, non, finalement c’était mieux dans un seul graphique, mais peux-tu ajouter une courbe de lissage par groupe pour voir si la tendance est la même?
ggplot(data = msleep) +
geom_point(mapping = aes(
x = sleep_rem,
y = awake,
color = vore
)) +
geom_smooth(mapping = aes(
x = sleep_rem,
y = awake,
color = vore
)
)
`geom_smooth()` using method = 'loess' and formula 'y ~ x'
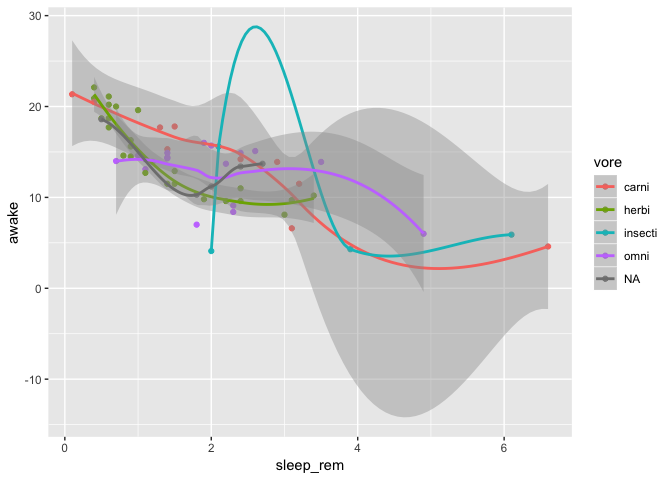
Ouin, c’est trop un fouilli, peux-tu me mettre juste des régressions linéaires finalement?
ggplot(data = msleep) +
geom_point(mapping = aes(
x = sleep_rem,
y = awake,
color = vore
)) +
geom_smooth(
mapping = aes(
x = sleep_rem,
y = awake,
color = vore
),
method = "lm",
se = FALSE
)
Warning: Removed 22 rows containing non-finite values (stat_smooth).
Warning: Removed 22 rows containing missing values (geom_point).
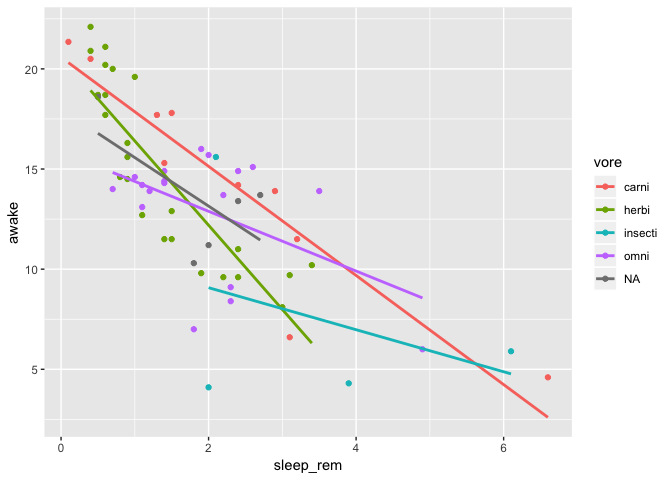
Pour éviter la duplication, les mappings globaux
Si vous voulez réutiliser des associations entre les propriétés graphiques et
vos données dans plusieurs couches, vous pouvez les mentionner lors de la
création de l’objet ggplot :
ggplot(data = msleep, mapping = aes(
x = sleep_rem,
y = awake,
color = vore
)) +
geom_point() +
geom_smooth(
method = "lm",
se = FALSE
)
Warning: Removed 22 rows containing non-finite values (stat_smooth).
Warning: Removed 22 rows containing missing values (geom_point).
Et pour condenser votre code encore plus
Vous pouvez profiter du fait que dans R, le nom des arguments est optionel, du
moment que vous respectez l’ordre spécifié dans la documentation (?ggplot):
Usage
ggplot(data = NULL, mapping = aes(), ..., environment = parent.frame())
Ce qui permet de faire :
ggplot(msleep, aes(
x = sleep_rem,
y = awake,
color = vore
)) +
geom_point() +
geom_smooth(
method = "lm",
se = FALSE
)
Warning: Removed 22 rows containing non-finite values (stat_smooth).
Warning: Removed 22 rows containing missing values (geom_point).
Quelles couches graphiques sont disponibles?
Une variable continue
ggplot(msleep) +
geom_histogram(aes(x = awake))
`stat_bin()` using `bins = 30`. Pick better value with `binwidth`.
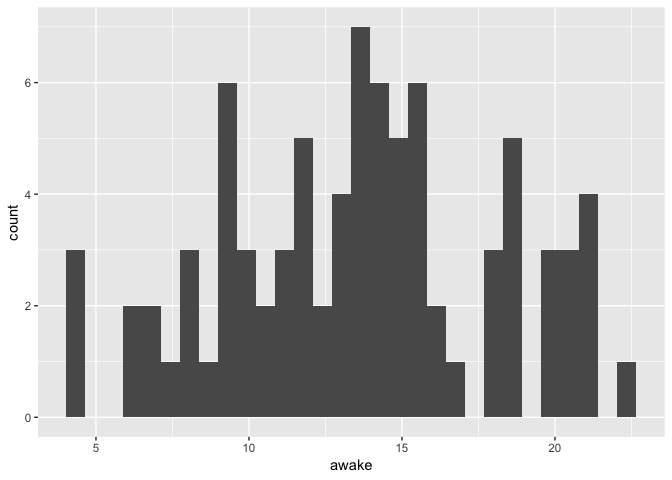 Prenez note de l’avertissement pour le nombre de bandes de l’histogramme
Prenez note de l’avertissement pour le nombre de bandes de l’histogramme
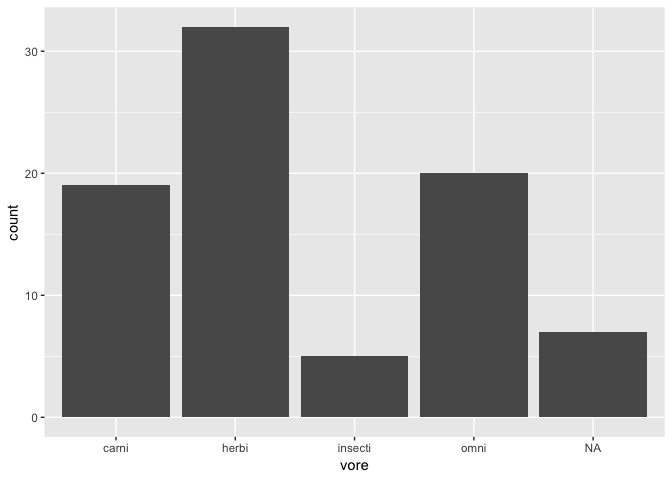
Une variable catégorique / discrète
ggplot(msleep) +
geom_bar(aes(x = vore))
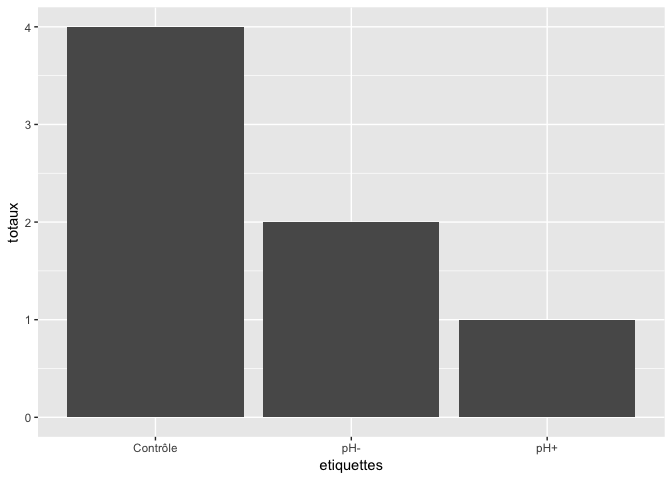
Attention, si vos valeurs sont déjà compilées, il faut plutôt passer par un autre geom, p. ex.
x <- data.frame(
totaux = c(4,1,2),
etiquettes = c("Contrôle", "pH+", "pH-")
)
ggplot(x) +
geom_col(aes(x = etiquettes, y = totaux))
Deux variables continues
ggplot( msleep) +
geom_point(aes(x = sleep_rem, y = awake))
Warning: Removed 22 rows containing missing values (geom_point).
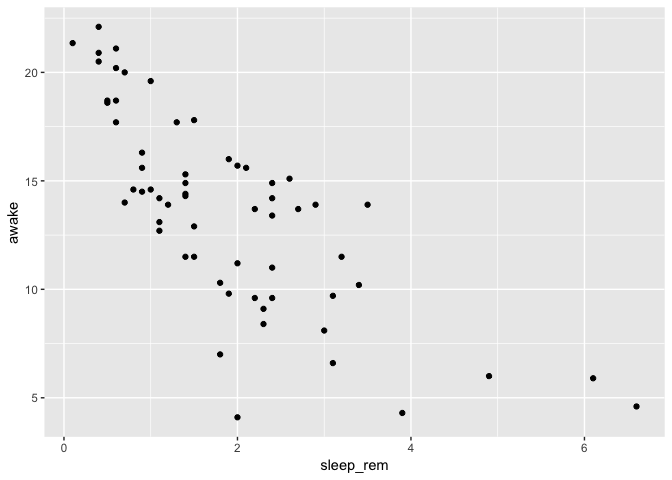
On peut aussi remplacer les points par du texte
ggplot(msleep) +
geom_text(aes(x = sleep_rem, y = awake, label = genus))
Warning: Removed 22 rows containing missing values (geom_text).
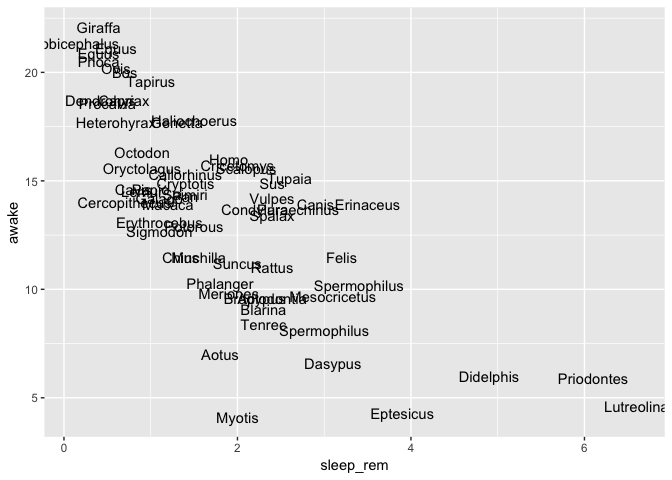
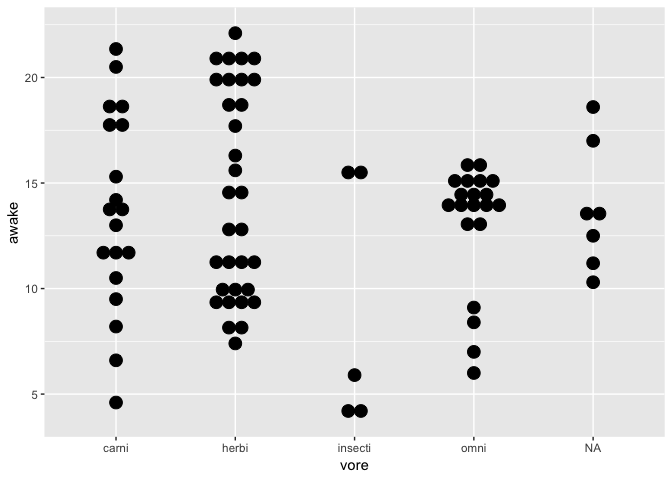
Une variable continue et une discrète
ggplot(msleep) +
geom_boxplot(aes(x = vore, y = awake))
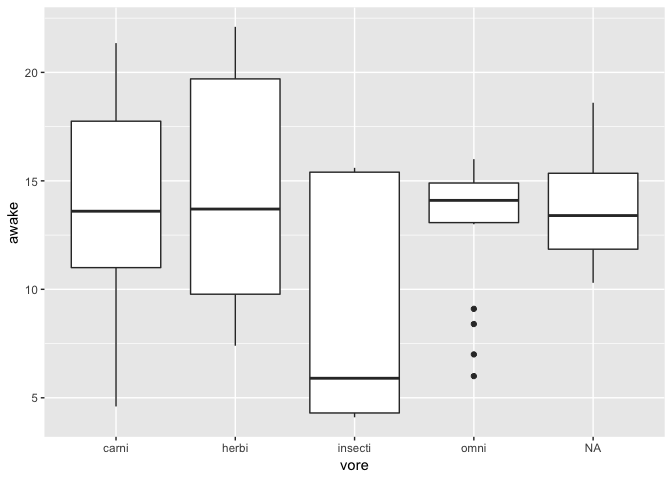
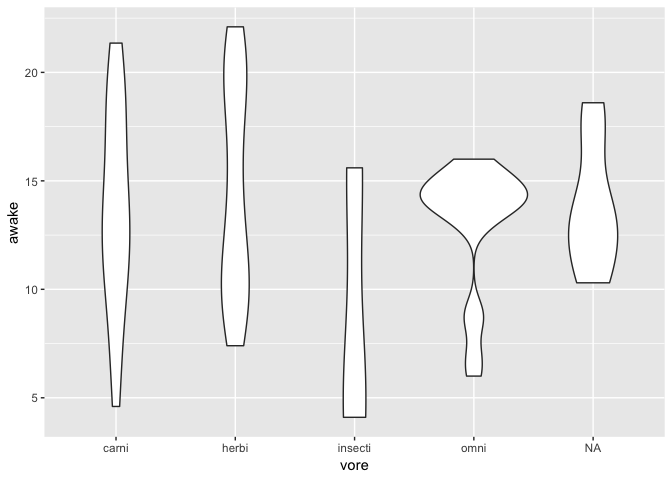
Ou de plus en plus utilisé, le violin plot
ggplot(msleep) +
geom_violin(
aes(x = vore, y = awake)
)
ggplot(msleep) +
geom_dotplot(
aes(x = vore, y = awake),
binaxis = "y",
stackdir = "center"
)
`stat_bindot()` using `bins = 30`. Pick better value with `binwidth`.
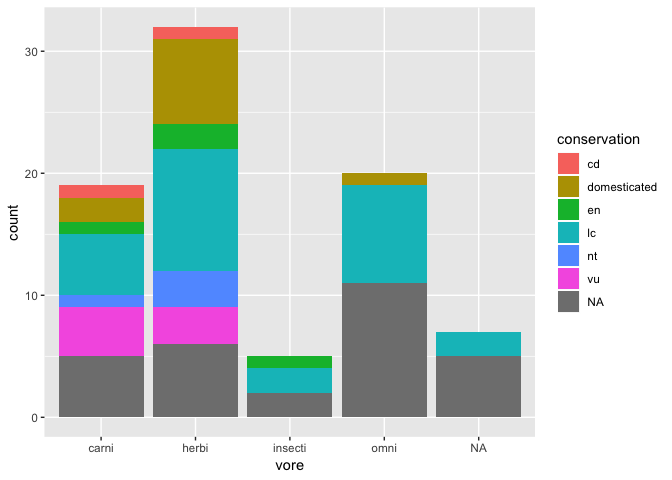
Deux variables discrètes
ggplot(msleep) +
geom_bar(aes(x = vore, fill = conservation))
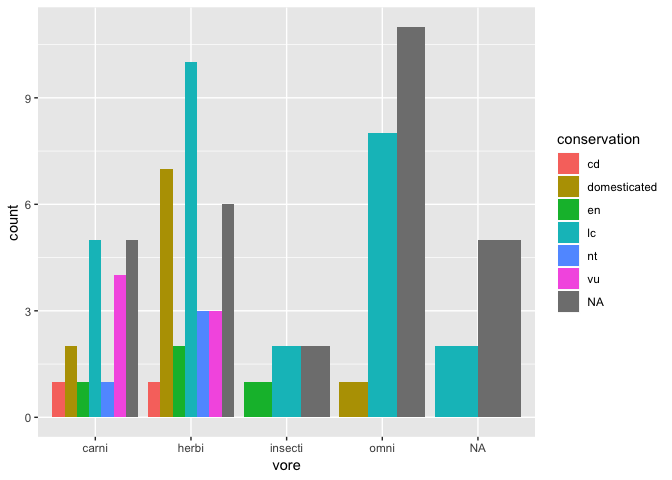
Modificateurs de position
On peut utiliser le modificateur de position pour organiser le diagramme à bandes d’autres façons
ggplot(msleep) +
geom_bar(
aes(x = vore, fill = conservation),
position = "dodge"
)
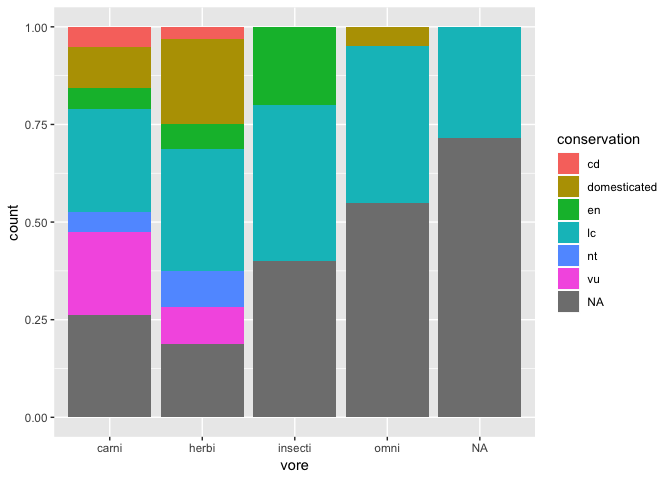
ggplot(msleep) +
geom_bar(
aes(x = vore, fill = conservation),
position = "fill"
)
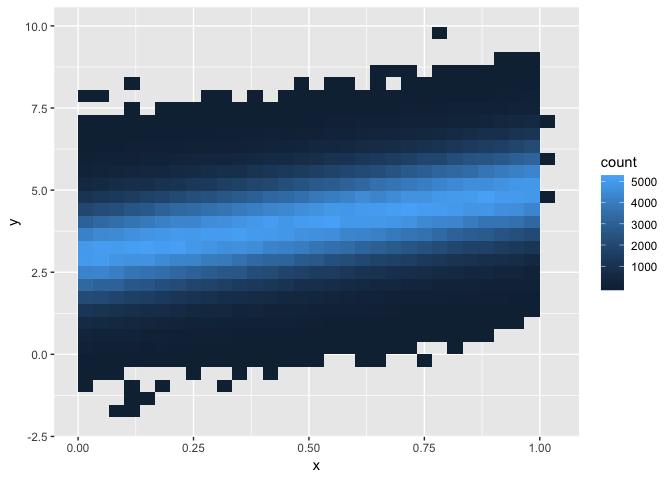
Si vous avez des millions de points
n <- 1000000
x <- runif(n)
y <- 3 + 2*x + rnorm(n)
df <- data.frame(
x = x,
y = y
)
ggplot(df,aes(x = x, y = y)) + geom_bin2d()
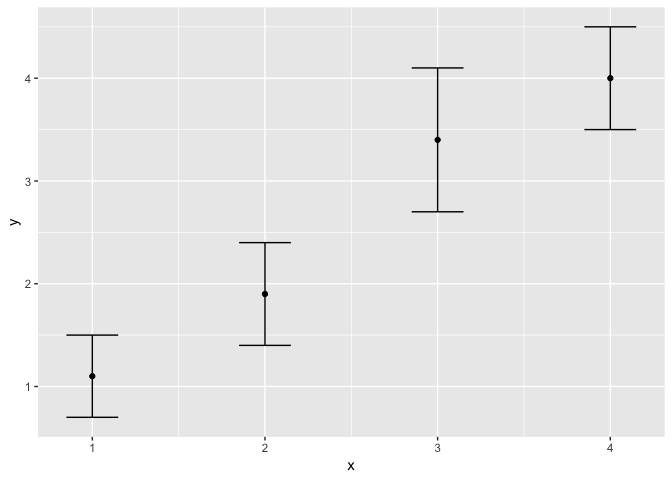
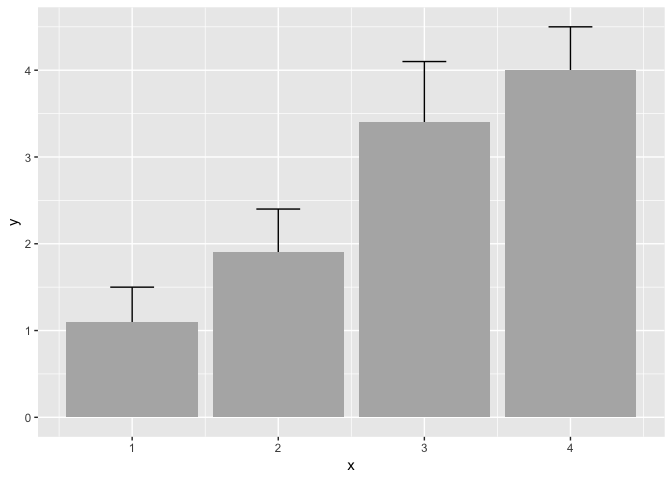
Visualisation de l’incertitude
df <- data.frame(
x = c(1,2,3,4),
y = c(1.1,1.9,3.4,4),
se = c(0.4, 0.5, 0.7, 0.5)
)
ggplot(df, aes(x = x, y = y)) +
geom_point() +
geom_errorbar(
aes(ymin = y - se, ymax = y + se),
width = 0.3
)
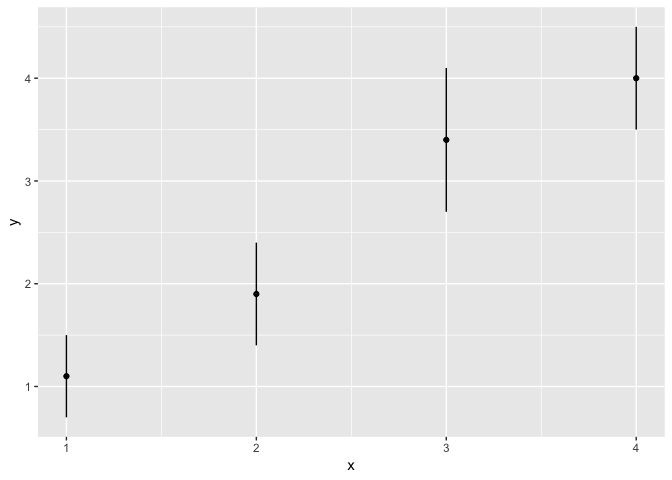
ggplot(df, aes(x = x, y = y)) +
geom_point() +
geom_linerange(
aes(ymin = y - se, ymax = y + se)
)
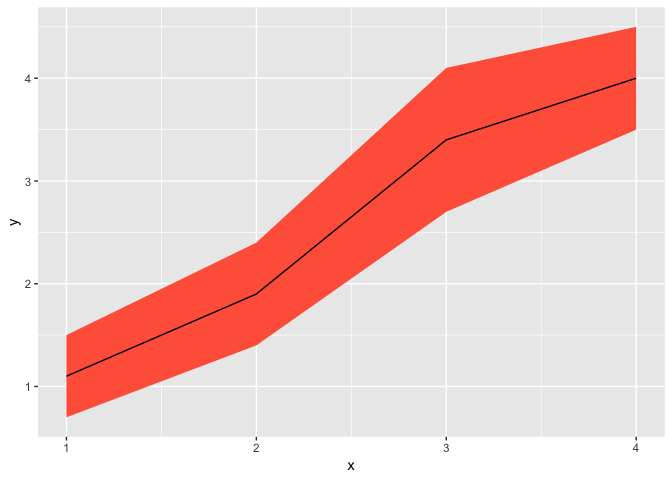
ggplot(df, aes(x = x, y = y)) +
geom_ribbon(
aes(ymin = y - se, ymax = y + se),
fill = "tomato"
) +
geom_line()
Exercice #2
À l’aide des outils vus dans les dernières sections, reproduisez cette façon classique (mais peu recommandable!) de visualiser la différence entre les groupes, à l’aide des données suivantes :
df <- data.frame(
x = c(1,2,3,4),
y = c(1.1,1.9,3.4,4),
se = c(0.4, 0.5, 0.7, 0.5)
)
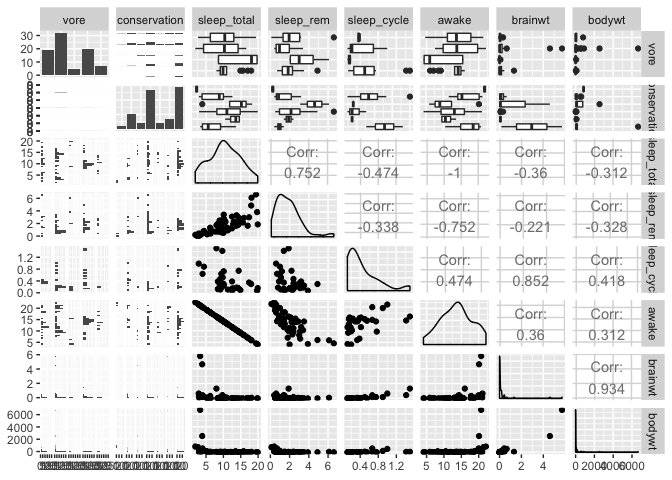
L’outil d’exploration ultime
library(GGally)
ggpairs(msleep[,-c(1,2,4)])
ggpairs(msleep[,-c(1,2,4)], aes(col = vore))

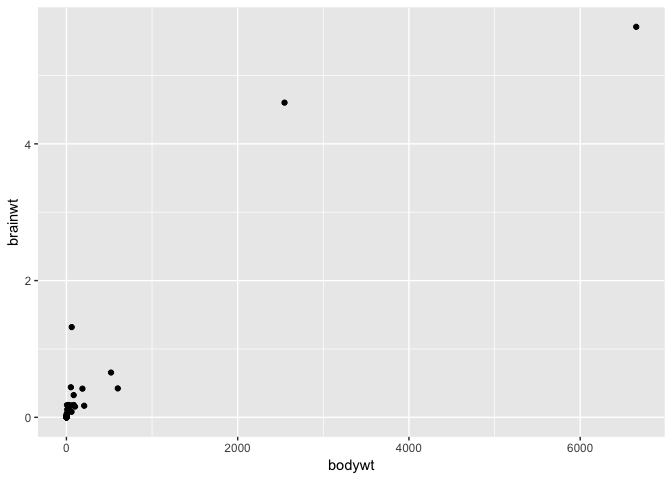
Transformations rapides (sans toucher aux données)
On peut ajouter une transformation des axes comme un élément supplémentaire du graphique…
ggplot(msleep) +
geom_point(aes(x = bodywt, y = brainwt))
Warning: Removed 27 rows containing missing values (geom_point).
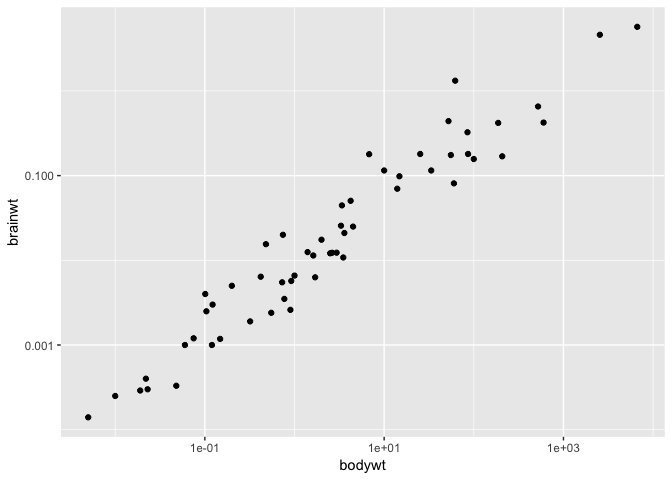
ggplot(msleep) +
geom_point(aes(x = bodywt, y = brainwt)) +
scale_x_log10() +
scale_y_log10()
Warning: Removed 27 rows containing missing values (geom_point).
Il existe aussi scale_x_reverse et scale_x_sqrt
Avant de fignoler, apprendre à bien sauvegarder…
La fonction ggsave permet d’envoyer dans un fichier le dernier graphique
produit
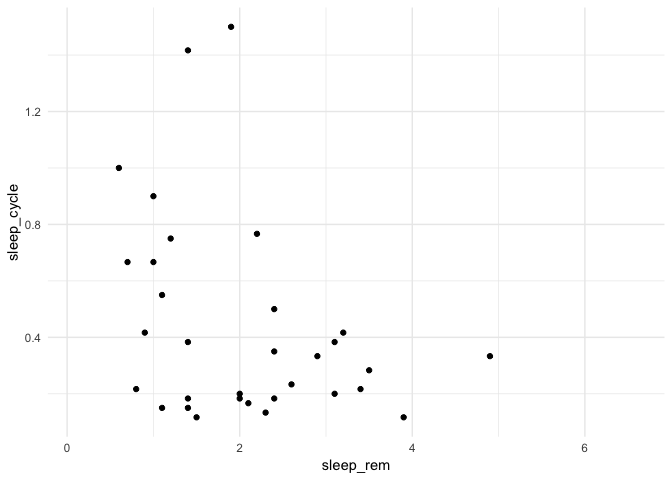
ggplot(msleep) +
geom_point(aes(x = sleep_rem, y = sleep_cycle))
ggsave(filename = "/assets/RapidDataViz_files/Resultats/Fig1.jpg")
Saving 7 x 5 in image
NB le nom du dossier dans lequel vous tentez d’écrire doit déjà exister…
Le type de fichier est choisi automatiquement à l’aide de l’extension choisie (pdf, jpg, png, eps, etc.)
Comment déterminer les dimensions?
Pas d’autres solutions que d’y aller par essai/erreur. ggplot change la taille relative des points, du texte etc. selon les dimensions désirées, vous devrez expérimenter.
ggsave(filename = "/assets/RapidDataViz_files/Resultats/2x2.jpg", width = 2, height = 2)
ggsave(filename = "/assets/RapidDataViz_files/Resultats/8x8.jpg", width = 8, height = 8)
Par défaut les unités sont en pouces, mais vous pouvez les changer pour de cm
avec l’argument units="cm"
Comment modifier la résolution
Pour les fichiers qui ne sont pas vectoriels (p. ex. jpg, png), vous pouvez aussi spécifier la qualité d’image, en nombre de points par pouces (dot per inches; dpi)
ggsave(filename = "/assets/RapidDataViz_files/Resultats/72.jpg", width = 2, height = 2, dpi = 72)
ggsave(filename = "/assets/RapidDataViz_files/Resultats/1200.jpg", width = 2, height = 2, dpi = 1200)
On recommande souvent 300 dpi pour des graphiques qui seront reproduits en version papier…
Et finalement, le fameux fond gris…
ggplot(msleep) +
geom_point(aes(x = sleep_rem, y = sleep_cycle)) +
theme_classic()
ggplot(msleep) +
geom_point(aes(x = sleep_rem, y = sleep_cycle)) +
theme_dark()
ggplot(msleep) +
geom_point(aes(x = sleep_rem, y = sleep_cycle)) +
theme_minimal()