ggplot2 avancé - problèmes et solutions
Charles Martin , édité par Jade Dormoy-Boulanger
Janvier 2024
- Faire ressortir de l’information
- Problèmes classiques en limnologie
- Problèmes classiques en écologie végétale
- Personnalisation
- Pour la publication
- Autres / Misc.
Cette formation assume que vous êtes déjà familières avec les fonctions de base de ggplot2. Si ce n’est pas le cas, prenez le temps de parcourir la formation de base (https://numerilab.io/fr/ateliers/RapidDataViz) avant de poursuivre.
L’atelier d’aujourd’hui est organisé comme un Problèmes et solutions. J’ai essayé de ramasser les problèmes rencontrés le plus souvent quand on confronte les connaissances de base de ggplot2 à la réalité des sciences de l’environnement.
J’ai essayé d’organiser le matériel par thème, mais ce n’est rien de miraculeux, mon atelier est quand même un gigantesque melting pot.
Faire ressortir de l’information
Annotations
Une chose qui peut nous arriver de temps à autres est de vouloir ajouter à un
graphique un petit bout d’information ne provenant pas directement de notre
tableau de données. On peut évidemment s’en sortir en trichant et en ajouter
ces données d’une façon ou d’une autre à nos données, mais les versions
récentes de ggplot2 incluent une fonction annotate, qui nous permet de
faire cela beaucoup plus proprement.
library(ggplot2)
ggplot(msleep, aes(x = sleep_total, y = sleep_rem)) +
geom_point() +
annotate("point", x = 15, y = 6, color = "red", size = 3) +
annotate("text", x = 15.5, y = 5.6, label = "Drôle de bibite")
Warning: Removed 22 rows containing missing values (`geom_point()`).
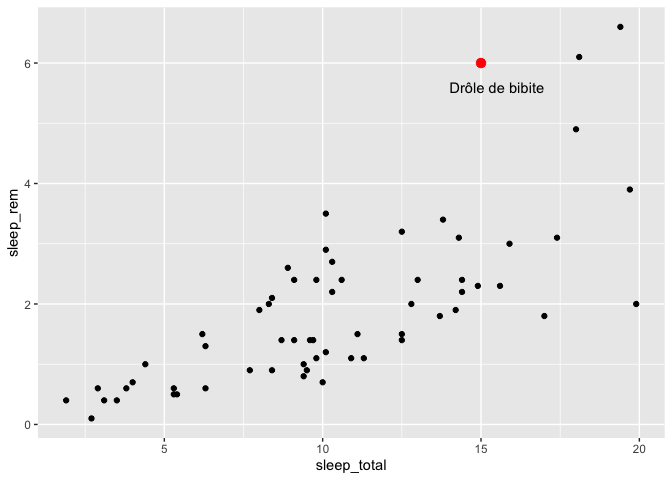
Vous avez ainsi accès à toute la palette de geoms.
Combiner des données de plusieurs sources dans un même graphique
Avant de vous lancer dans du copier-coller pour ajouter une série d’annotations, une chose intéressante à savoir est que l’on peut spéficier que certaines couches de points proviennent d’un tableau de données différent de celui passé à la fonction ggplot.
On pourrait par exemple se préparer un tableau avec les moyennes par type d’alimentation :
library(dplyr)
Attaching package: 'dplyr'
The following objects are masked from 'package:stats':
filter, lag
The following objects are masked from 'package:base':
intersect, setdiff, setequal, union
moyennes <- msleep %>%
group_by(vore) %>%
summarize(
sleep_total = mean(sleep_total, na.rm = TRUE),
sleep_rem = mean(sleep_rem, na.rm = TRUE)
)
moyennes
# A tibble: 5 × 3
vore sleep_total sleep_rem
<chr> <dbl> <dbl>
1 carni 10.4 2.29
2 herbi 9.51 1.37
3 insecti 14.9 3.52
4 omni 10.9 1.96
5 <NA> 10.2 1.88
(remarquez que j’utilise na.rm pour permettre le calcul rapidement malgré les NA. Dans la vraie vie, il aurait fallu nettoyer correctement le tableau de données avant d’arriver ici…)
Pour se faciliter la vie, c’est une bonne idée de garder le même nom de variables comme j’ai fait ci-haut. Mais sinon, on pourrait simplement associer nos nouveaux noms avec un aes() dans ce geom…
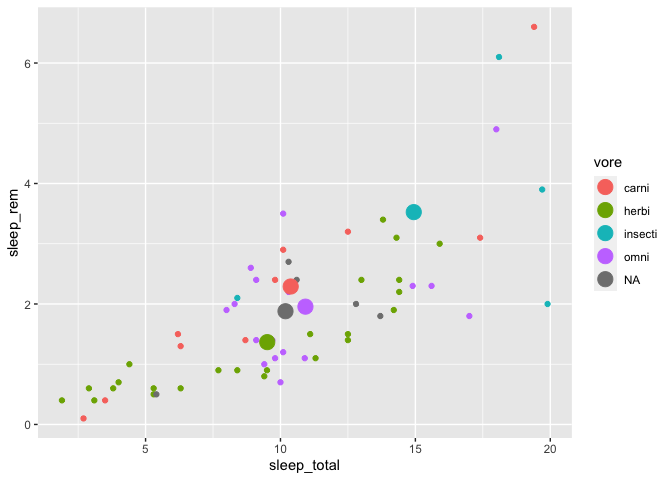
ggplot(msleep, aes(x = sleep_total, y = sleep_rem, color = vore)) +
geom_point() +
geom_point(data = moyennes, size = 5)
Warning: Removed 22 rows containing missing values (`geom_point()`).
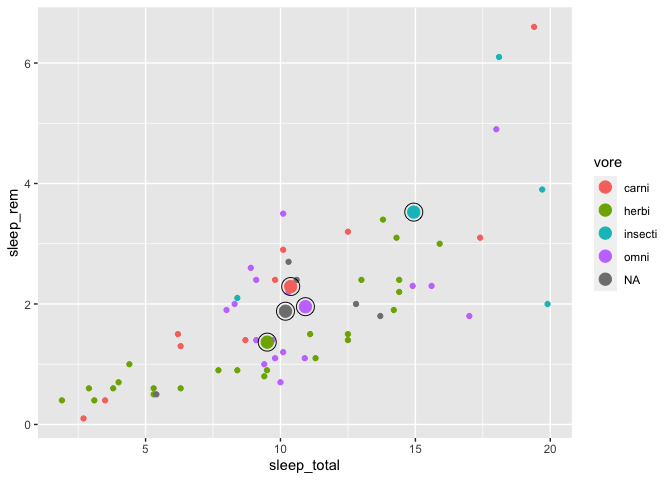
Et un petit truc pour attirer l’attention sur certains points, une des choses que j’aime bien faire est d’ajouter un cercle vide un peu plus grand autour de chaque point.
ggplot(msleep, aes(x = sleep_total, y = sleep_rem, color = vore)) +
geom_point() +
geom_point(data = moyennes, size = 4) +
geom_point(data = moyennes, size = 6, shape = 1, color = "black")
Warning: Removed 22 rows containing missing values (`geom_point()`).
Problèmes classiques en limnologie
Jouer avec les axes
Le premier problème concret que nous verrons est : comment intégrer un second axe des Y à droite dans un graphique de ggplot2. Il s’agit en principe de quelque chose de simple, que l’on voit souvent en limnologie où l’on met par exemple sur un même graphique le pH et la température de l’eau.
Ce genre de choses n’est pas simple dans ggplot2, parce que l’auteur du package (Hadley Wickham) déteste cette façon de faire, entre autres parce qu’elle peut être facilement manipulée pour tromper le lecteur (https://stackoverflow.com/questions/3099219/ggplot-with-2-y-axes-on-each-side-and-different-scales/3101876#3101876).
Il a donc (malheureusement?) fait exprès que ce soit complexe d’ajouter un deuxième axe des Y, pour éviter que les gens s’en servent à la légère.
Quoiqu’il en soit, voici un petit jeu de données limnologie-style pour explorer comment nous pouvons faire pour mettre un deuxième axe des Y.
limno <- data.frame(
Profondeur = c(1,2,3,4),
O2 = c(90, 75, 70,68),
Temperature = c(12.1,10.8,10.4,8.5)
)
ggplot(limno, aes(x = Profondeur)) +
geom_line(aes(y = O2))
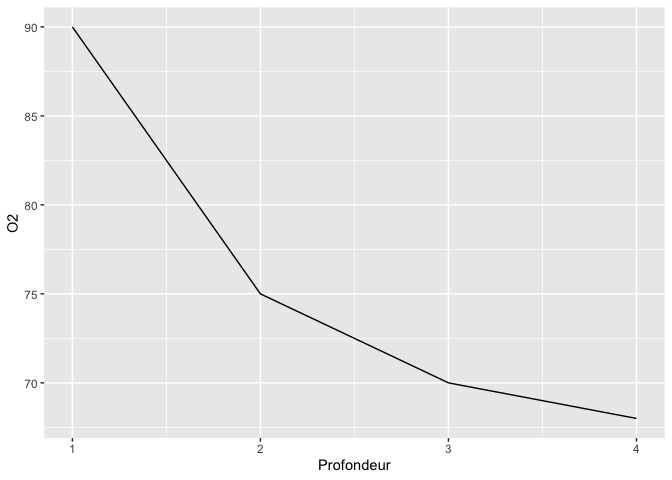
Alors, la première étape est d’ajouter notre deuxième série de données, ici la température :
ggplot(limno, aes(x = Profondeur)) +
geom_line(aes(y = O2), color = "blue", linetype = "dashed") +
geom_line(aes(y = Temperature), color = "red")
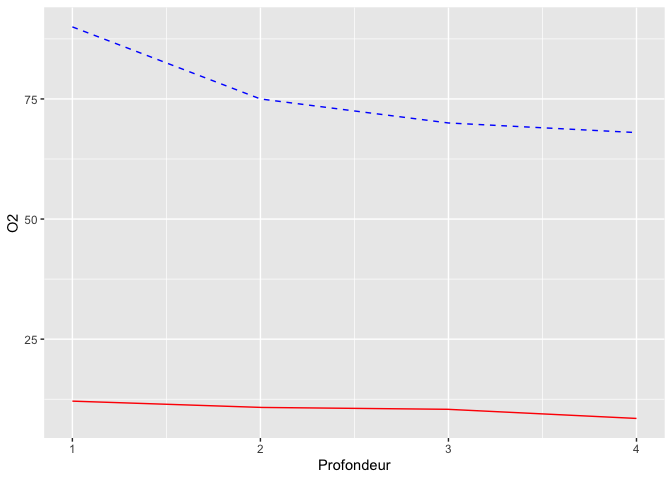
Remarquez que je modifie les propriétés des lignes à l’extérieur du “aes”, puisque cette information est identique pour chaque ligne du tableau.
Donc on a maintenant nos deux séries de chiffres, mais notre graphique ne sait pas qu’il faut mettre une autre échelle pour la température.
L’étape suivante est de se demander quelle transformation il faudrait appliquer pour ramener le maximum de température à la même hauteur que celui d’oxygène. Une façon rapide est de calculer le rapport du maximum entre nos deux variables : 90/12 = 7.5. La température doit donc être multipliée par 7.5
ggplot(limno, aes(x = Profondeur)) +
geom_line(aes(y = O2), color = "blue", linetype = "dashed") +
geom_line(aes(y = Temperature*7.5), color = "red")
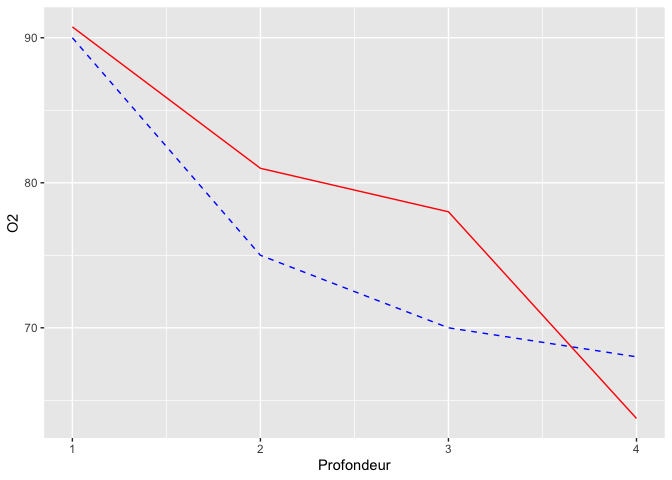
Maintenant que les données sont ajustées, il faut appliquer cette transformation à l’inverse à l’axe pour terminer l’ajustement :
ggplot(limno, aes(x = Profondeur)) +
geom_line(aes(y = O2), color = "blue", linetype = "dashed") +
geom_line(aes(y = Temperature*7.5), color = "red") +
scale_y_continuous(sec.axis = sec_axis(~./7.5,name = "Température"))
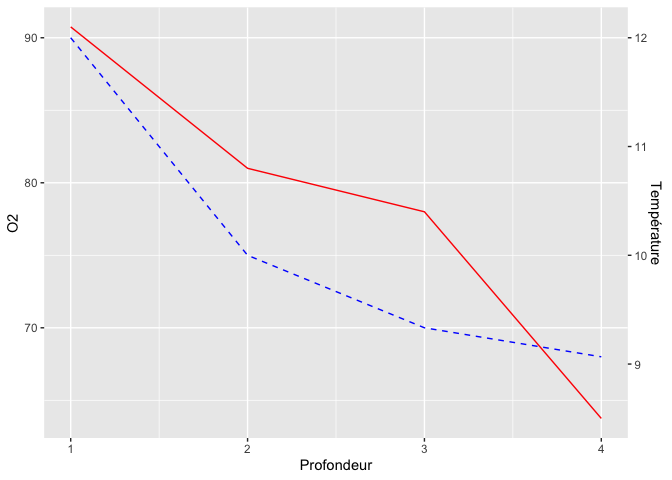
Dans ce genre de graphique, il arrive aussi fréquemment que l’on veuille “flipper” les axes, pour mettre la profondeur en Y et nos deux autres mesures en X, pour imiter l’organisation spatiale dans un lac. Il existe pour cela la fonction coord_flip :
ggplot(limno, aes(x = Profondeur)) +
geom_line(aes(y = O2), color = "blue", linetype = "dashed") +
geom_line(aes(y = Temperature*7.5), color = "red") +
scale_y_continuous(sec.axis = sec_axis(~./7.5,name = "Température")) +
coord_flip()
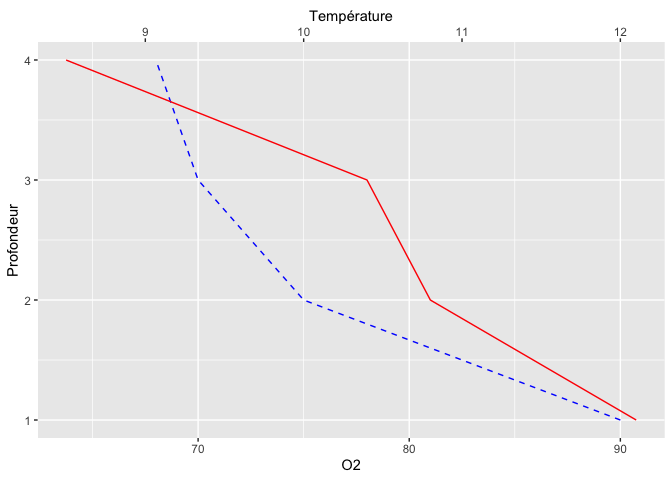
Remarquez que naturellement, si on veut vraiment imiter le lac, l’axe des X (présenté à la verticale) devrait aussi être inversé, avec la valeur de zéro en haut. On peut inverser un axe, comme ceci :
ggplot(limno, aes(x = Profondeur)) +
geom_line(aes(y = O2), color = "blue", linetype = "dashed") +
geom_line(aes(y = Temperature*7.5), color = "red") +
scale_y_continuous(sec.axis = sec_axis(~./7.5,name = "Température")) +
coord_flip() +
scale_x_reverse()
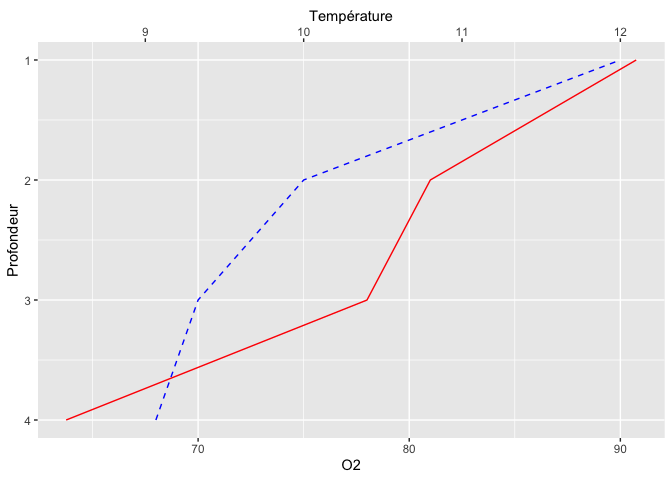
Les expressions
Dernière chose tannante avec ce graphique, c’est que le symbole pour l’oxygène devrait avoir le 2 en indice, plus petit qu’un 2 ordinaire. On peut, pour se faire, utiliser la fonction expression qui nous permet d’écrire une formule mathématique, dans un langage ressemblant un peu au Latex.
On peut en profiter pour changer le thème en finalisant ce graphique.
ggplot(limno, aes(x = Profondeur)) +
geom_line(aes(y = O2), color = "blue", linetype = "dashed") +
geom_line(aes(y = Temperature*7.5), color = "red") +
scale_y_continuous(sec.axis = sec_axis(~./7.5,name = "Température")) +
coord_flip() +
scale_x_reverse() +
labs(y=expression(O[2]))+
theme_minimal()
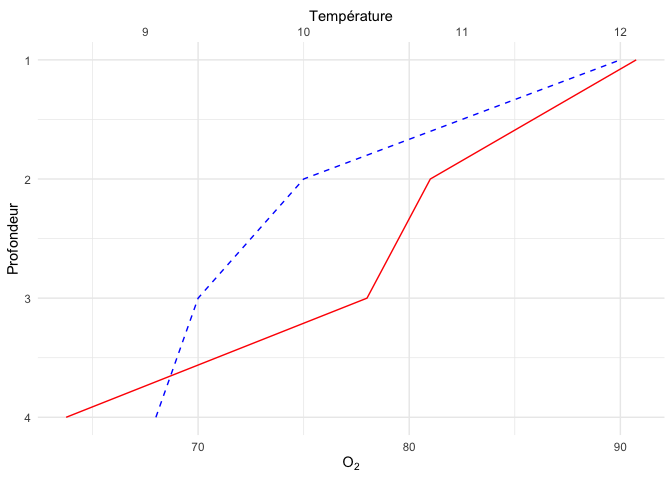
L’ensemble de la syntaxe pour écrire des expressions est défini dans cette page d’aide : https://stat.ethz.ch/R-manual/R-devel/library/grDevices/html/plotmath.html
Si on voulait par exemple écrire l’équation de la variance d’une population, le guide nous indique d’écrire ceci :
msleep %>%
ggplot() +
annotate("text",x=1,y=1,size = 10,label=expression(
sigma^2 = sum(
over(
(x[i]-bar(x))^2,
n
),
i==1,
n)
))
Error: <text>:4:13: unexpected '='
3: annotate("text",x=1,y=1,size = 10,label=expression(
4: sigma^2 =
^
Et là, évidemment, ça ne fonctionne pas! Pourtant, on respecte tout ce qui écrit dans le guide Charles…
La nuance importante à comprendre est que le code utilisé pour écrire l’expression doit être du code qui respecte aussi la syntaxe de R. On n’aurait jamais eu le droit d’écrire x^2=3 dans R.
La solution dans ce cas là est d’utiliser la fonction paste pour coller ensemble des morceaux qui sont valides individuellement, comme ceci :
msleep %>%
ggplot() +
annotate("text",x=1,y=1,size = 10,label=
expression(paste(
sigma^2,
"=",
sum(over((x[i]-bar(x))^2,n),i==1,n)
))
)
Warning in is.na(x): is.na() applied to non-(list or vector) of type
'expression'
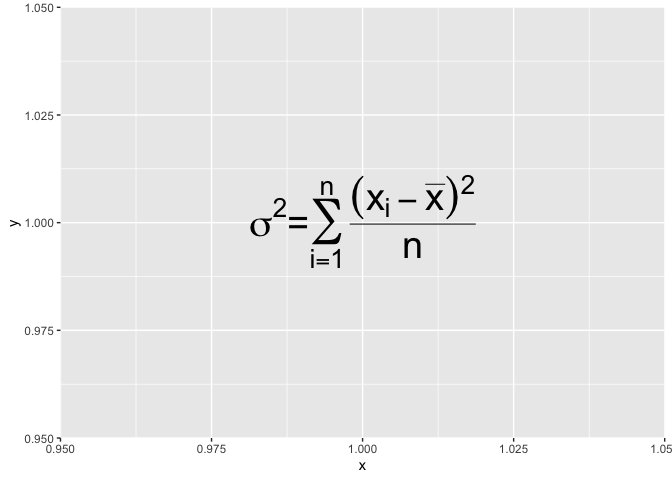
Une fois l’idée du paste illustrée, remarquez que la vraie façon d’indiquer un “=” dans le guide est d’utiliser “==” ;-)
msleep %>%
ggplot() +
annotate("text",x=1,y=1,size = 10,label=
expression(paste(
sigma^2 == sum(over((x[i]-bar(x))^2,n),i==1,n)
))
)
Warning in is.na(x): is.na() applied to non-(list or vector) of type
'expression'
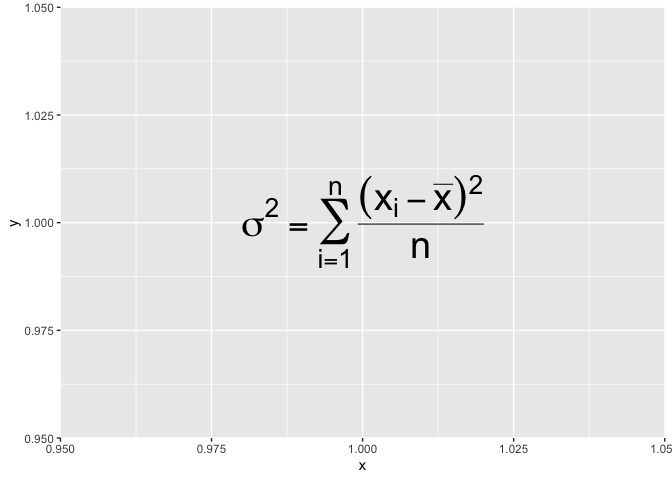
Problèmes classiques en écologie végétale
Équation de régression
Un autre cas où nous avons souvent besoin d’ajouter des annotations à un graphique est lorsque nous traçons une régression.
La chose à savoir, c’est qu’il n’y a pas de façon simple d’ajouter l’équation en utilisant geom_smooth(method = "lm"). Cette méthode est faite pour explorer les données et non pas pour ajuster des modèles et en extraire les paramètres. La méthode que je vous montre ici vous sera utile dans beaucoup plus de situations qu’uniquement la régression linéaire.
Elle consiste à ajuster le modèle, pour ensuite ajouter les prédictions du modèle au tableau de données pour pouvoir tracer la droite.
library(tidyr)
bd <- msleep %>%
select(sleep_total, sleep_rem) %>%
drop_na
m <- lm(sleep_rem ~ sleep_total, data = bd)
bd <- bd %>%
mutate(
prediction = predict(m)
)
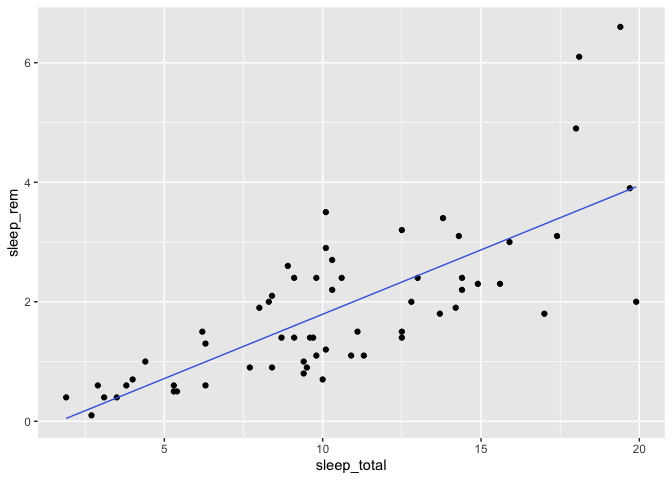
bd %>%
ggplot(aes(x = sleep_total, y = sleep_rem)) +
geom_point() +
geom_line(aes(y = prediction), color = "royalblue")
Pour ajouter l’équation, il faut d’abord trouver nos paramètres :
summary(m)
Call:
lm(formula = sleep_rem ~ sleep_total, data = bd)
Residuals:
Min 1Q Median 3Q Max
-1.9233 -0.5473 -0.1200 0.4791 2.7843
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) -0.36132 0.27833 -1.298 0.199
sleep_total 0.21531 0.02459 8.756 2.92e-12 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 0.8634 on 59 degrees of freedom
Multiple R-squared: 0.5651, Adjusted R-squared: 0.5578
F-statistic: 76.67 on 1 and 59 DF, p-value: 2.916e-12
Puis on peut les ajouter avec une annotation de texte, que l’on peut combiner à la fonction expression si on veut être fancy :
bd %>%
ggplot(aes(x = sleep_total, y = sleep_rem)) +
geom_point() +
geom_line(aes(y = prediction), color = "royalblue") +
annotate("text",hjust = "left", x =3, y = 6, label = expression(
y[i] == -0.36 + 0.22*x[i]+epsilon[i]
), size = 8) +
annotate("text",hjust = "left",x =3, y = 5, label = expression(r^2 == 0.56), size = 8)
Warning in is.na(x): is.na() applied to non-(list or vector) of type
'expression'
Warning in is.na(x): is.na() applied to non-(list or vector) of type
'expression'
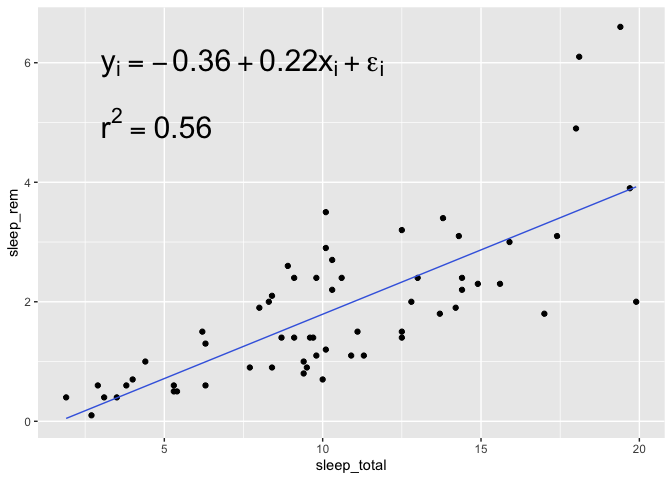
Remarquez que je modifie l’alignement horizontal du geom_text en utilisant “left” plutôt que “center” par défaut, ce qui permet d’aligner plus facilement nos deux annotations à gauche.
Densités dans l’espace
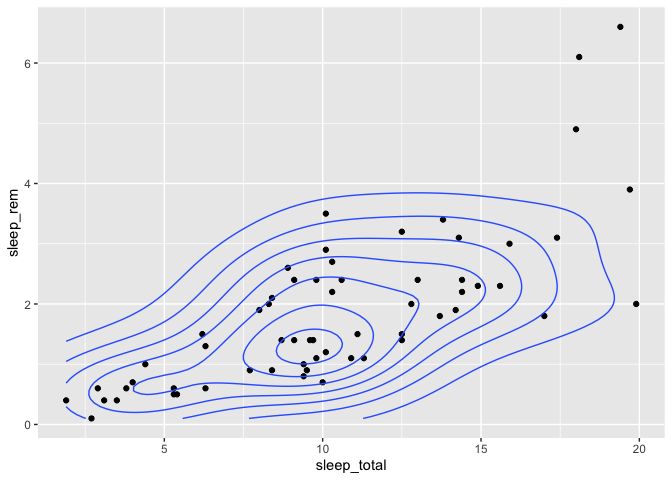
Si jamais vous avez récolté les coordonnées d’une série d’individus et que vous vouliez tracer une carte des densités dans l’espace pour trouver des patrons, il existe une couche exprès pour cela dans ggplot2, qui se nomme geom_density_2d.
Pour illustrer la fonction, nous allons utiliser sleep_rem et sleep_total comme variables X et Y, mais le principe est exactement le même :
msleep %>%
ggplot(aes(x = sleep_total, y = sleep_rem)) +
geom_point() +
geom_density_2d()
Warning: Removed 22 rows containing non-finite values (`stat_density2d()`).
Warning: Removed 22 rows containing missing values (`geom_point()`).
Vous pouvez aussi choisir un remplissage plutôt que des lignes (ou une combinaison des deux avec les deux couches)
msleep %>%
ggplot(aes(x = sleep_total, y = sleep_rem)) +
geom_density_2d_filled() +
geom_density_2d(color = "black") +
geom_point()
Warning: Removed 22 rows containing non-finite values
(`stat_density2d_filled()`).
Warning: Removed 22 rows containing non-finite values (`stat_density2d()`).
Warning: Removed 22 rows containing missing values (`geom_point()`).
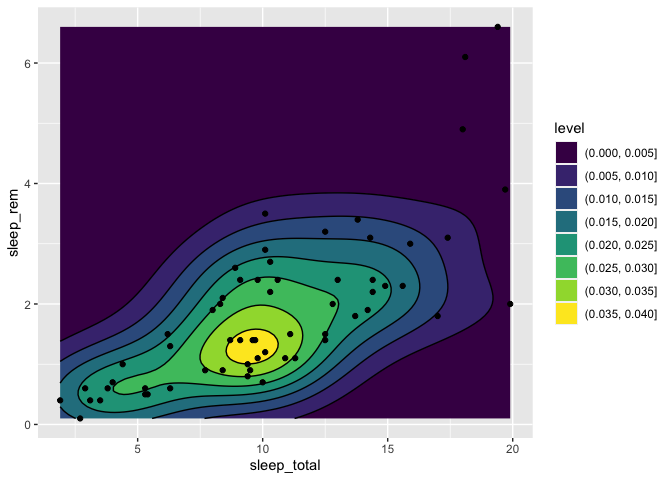
Personnalisation
Maintenant, laissons un peu la limnologie et l’écologie végétale derrière nous, et attaquons-nous à quelques trucs pour personnaliser nos graphiques.
Choisir manuellement les couleurs d’une échelle discrète
Une des critique qui revient souvent à propos des ggplot2 est le choix des couleurs, qui ne sont pas très sérieuses aux yeux de certains.
Il existe deux façons de modifier une échelle de couleur. La première consiste à simplement choisir une nouvelle palette, à partir d’une liste pré-établie. Cette liste provient du package RColorBrewer et peut être consultée ici : https://www.r-graph-gallery.com/38-rcolorbrewers-palettes.html
Voyons comment on pourrait par exemple utiliser la palette Pastel1 dans un graphique :
ggplot(msleep, aes(x = sleep_total, y = sleep_rem)) +
geom_point(aes(color = vore)) +
scale_color_brewer(palette = "Pastel1")
Warning: Removed 27 rows containing missing values (`geom_point()`).
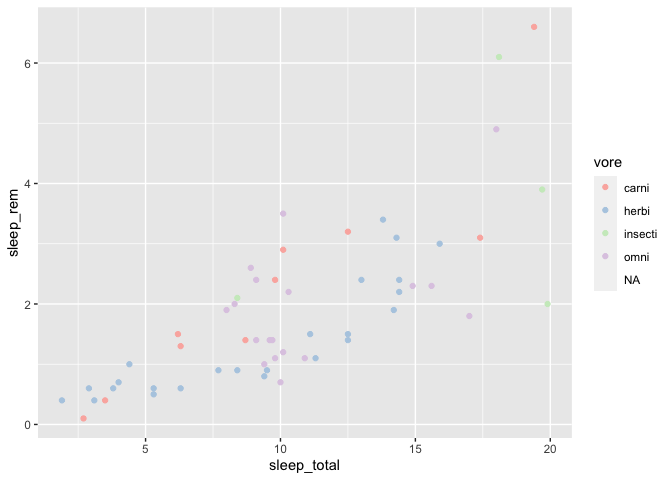
On aurait pu aussi définir les couleurs de notre palette manuellement, une par une. Pour se faire, il existe deux façons de faire, soit avec le nom des couleurs :
ggplot(msleep, aes(x = sleep_total, y = sleep_rem)) +
geom_point(aes(color = vore)) +
scale_color_manual(values = c(
"carni" = "red",
"herbi" = "green",
"insecti" = "yellow",
"omni" = "blue"
))
Warning: Removed 22 rows containing missing values (`geom_point()`).
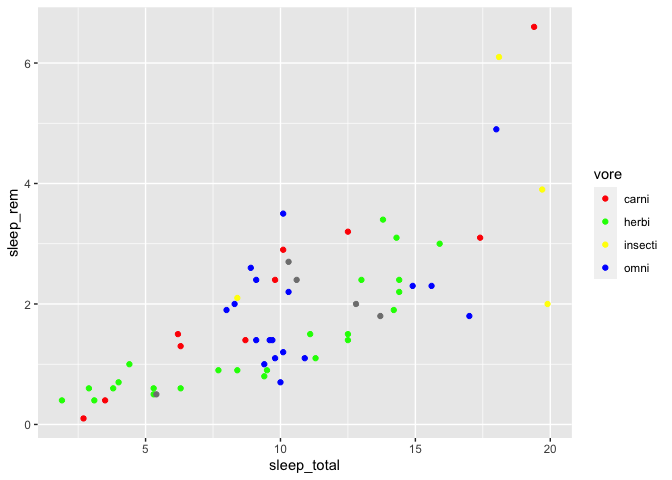
Consultez ce pdf pour la liste de toutes les couleurs ayant un nom dans R : http://www.stat.columbia.edu/~tzheng/files/Rcolor.pdf
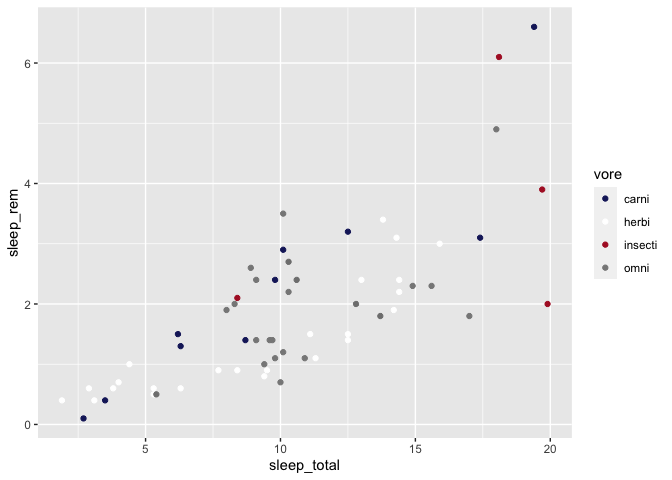
On aurait pu aussi construire manuellement nos couleurs à l’aide d’un code hexadécimal définissant la quantité de rouge, de bleu et de vert dans chaque couleur. Cela est très pratique pour copier par exemple une couleur précise retrouvée sur internet.
On aurait pu par exemple mettre notre graphique aux couleurs des Canadiens (https://teamcolorcodes.com/montreal-canadiens-color-codes/)
ggplot(msleep, aes(x = sleep_total, y = sleep_rem)) +
geom_point(aes(color = vore)) +
scale_color_manual(values = c(
"carni" = "#192168", # bleu
"herbi" = "#ffffff", # blanc
"insecti" = "#AF1E2D", # rouge
"omni" = "#888888" # gris?
))
Warning: Removed 22 rows containing missing values (`geom_point()`).
Définir un gradient de couleur
Lorsque la couleur associée à nos points doit représenter un variable quantitative plutôt que qualitative, on peut définir manuellement le gradient de couleurs qui sera utilisé par R.
Voyons d’abord un graphique avec le gradient de couleurs par défaut de R :
ggplot(msleep, aes(x = sleep_total, y = sleep_rem)) +
geom_point(aes(color = brainwt/bodywt))
Warning: Removed 22 rows containing missing values (`geom_point()`).
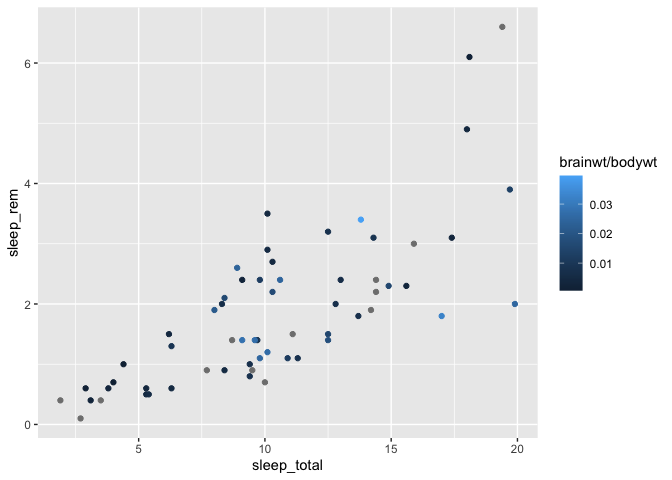
On peut modifier l’échelle de couleur utilisée, avec la fonction scale_color_gradient :
ggplot(msleep, aes(x = sleep_total, y = sleep_rem)) +
geom_point(aes(color = brainwt/bodywt)) +
scale_color_gradient(low = "blue", high= "red")
Warning: Removed 22 rows containing missing values (`geom_point()`).
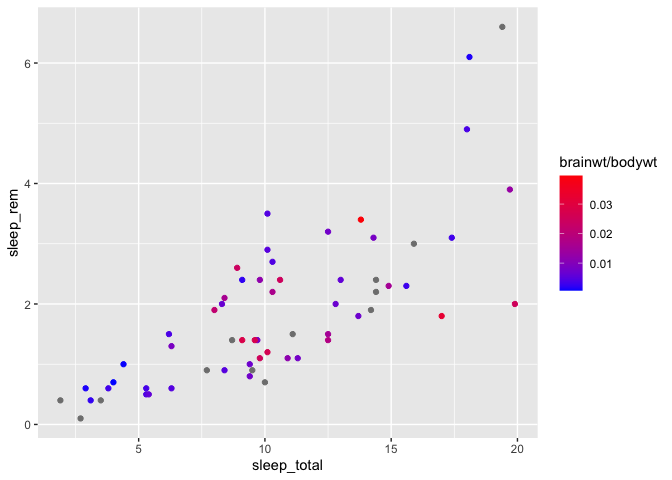
Si on veut choisir quelle sera la couleur intermédiaire du gradient, on peut utiliser une seconde fonction, scale_color_gradient2 :
ggplot(msleep, aes(x = sleep_total, y = sleep_rem)) +
geom_point(aes(color = brainwt/bodywt)) +
scale_color_gradient2(low = "blue", high= "red", mid = "white", midpoint = 0.02)
Warning: Removed 22 rows containing missing values (`geom_point()`).
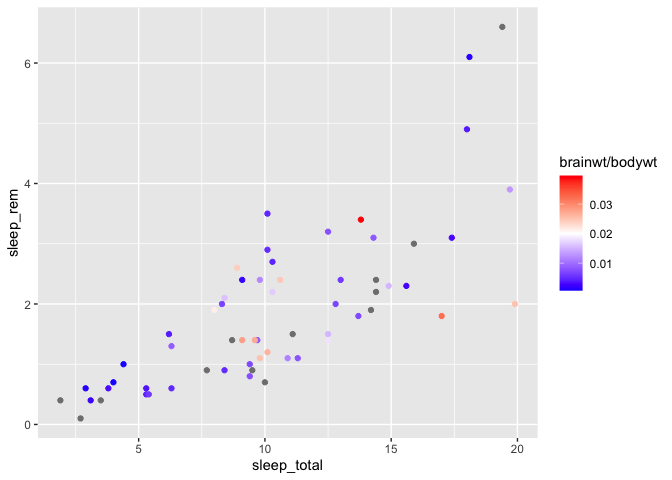
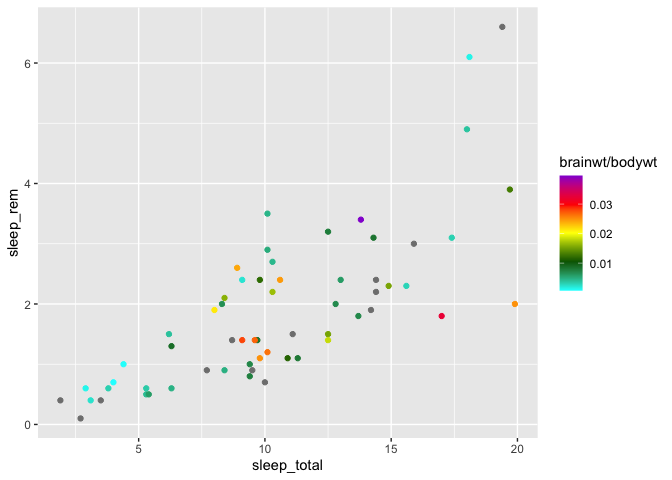
Si vous ne spécifiez pas de valeur pour le point milieu de votre gradient, ggplot assume que ce sera la valeur 0.
Vous pouvez aussi utiliser un nombre arbitraire de couleurs dans votre palette avec scale_color_gradientn. Par exemple, pour refaire (approximativement) la palette de couleur du radar d’Environnement Canada :
ggplot(msleep, aes(x = sleep_total, y = sleep_rem)) +
geom_point(aes(color = brainwt/bodywt)) +
scale_color_gradientn(
colors = c("cyan","darkgreen","yellow","red","darkviolet")
)
Warning: Removed 22 rows containing missing values (`geom_point()`).
Remarquez que toutes ces fonctions sont aussi disponibles pour les couleurs de remplissage. Remplacez simplement color pour fill, par exemple avec scale_fill_gradient2
Remarquez qu’ici aussi, on peut spécifier les couleurs avec un code hexadécimal. Par exemple, pour se faire un gradient tricolore :
ggplot(msleep, aes(x = sleep_total, y = sleep_rem)) +
geom_point(aes(color = brainwt/bodywt)) +
scale_color_gradient2(
high = "#192168",
mid = "#ffffff",
low = "#af1e2d", # remarquez que l'on peut utiliser majuscules ou minuscules ici, sans problèmes
midpoint = 0.02
)
Warning: Removed 22 rows containing missing values (`geom_point()`).
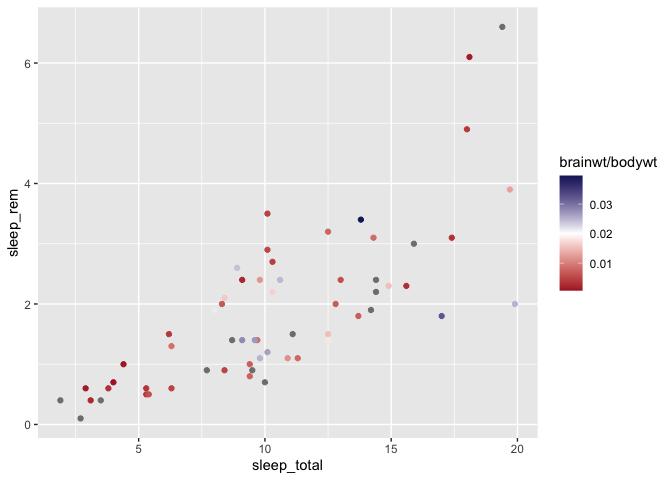
Il est à noter qu’il existe également des palettes spécifiquement pour les gens daltonniens, comme dans les libraries viridis, colorBlindness et colorblindr.
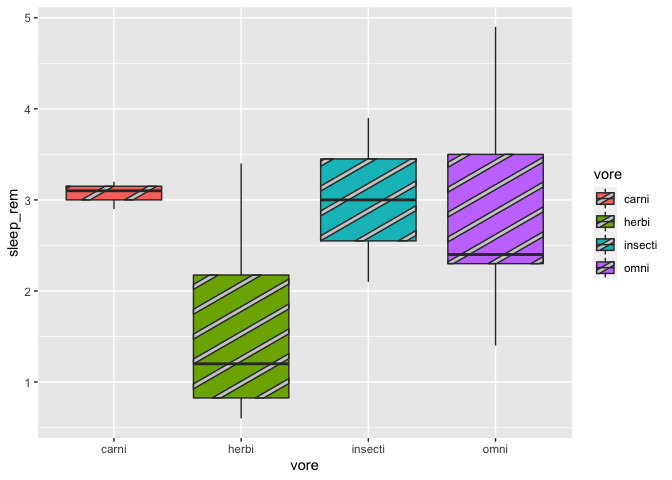
Finalement, certains pourraient aussi vouloir ajouter des motifs en plus des couleurs, pour des raisons esthétiques ou encore pour faciliter la lecture des graphiques des gens daltoniens. Il est donc possible de le faire grâce à la librarie ggpattern. Voici un exemple avec un diagramme à moustaches (boxplot). À noter que pour des raison de rapidité, on va simplement enlever les NAs du jeux de données, mais dans un contexte de recherche, il aurait fallu les adresser:
msleep2 <- na.omit(msleep)
library(ggpattern)
ggplot(msleep2, aes(x = vore, y = sleep_rem, fill = vore)) +
geom_boxplot_pattern(stat = "boxplot")
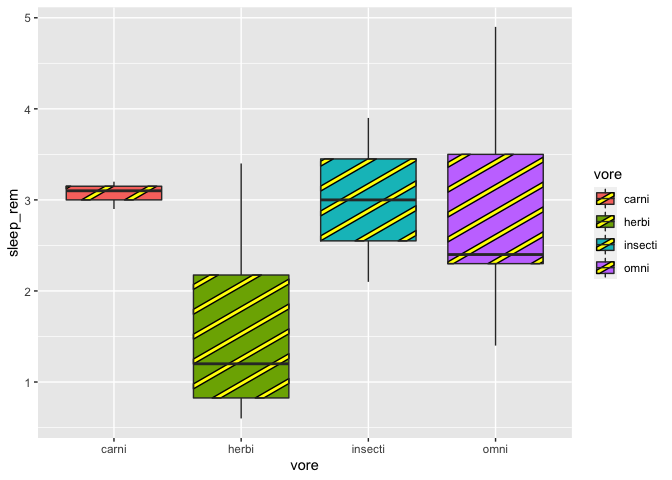
On peut également spécifier les couleurs des motifs comme ceci:
ggplot(msleep2, aes(x = vore, y = sleep_rem, fill = vore)) +
geom_boxplot_pattern(stat = "boxplot",
pattern_color = "black",
pattern_fill = "yellow")
Finalement, on peut changer les motifs en fonction des groupes représentés. Attention, il peut y avoir une erreur avec geom_boxplot_pattern (ggpattern est reelativement nouveau).Il faut lui spécifier les valeurs du motif, pour éviter qu’ils ne se répètent :
#avec l'erreur de motif
ggplot(msleep2, aes(x = vore, y = sleep_rem, fill = vore)) +
geom_boxplot_pattern(stat = "boxplot",
pattern_color = "black",
pattern_fill = "yellow",
aes(pattern = vore))
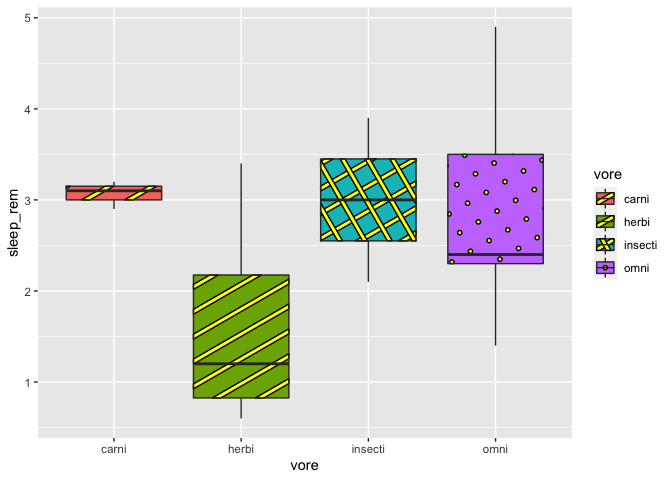
# avec la correction de l'erreur de motif
ggplot(msleep2, aes(x = vore, y = sleep_rem, fill = vore)) +
geom_boxplot_pattern(stat = "boxplot",
pattern = c("none", "stripe","crosshatch","circle"),
pattern_color = "black",
pattern_fill = "yellow",
show.legend = F)
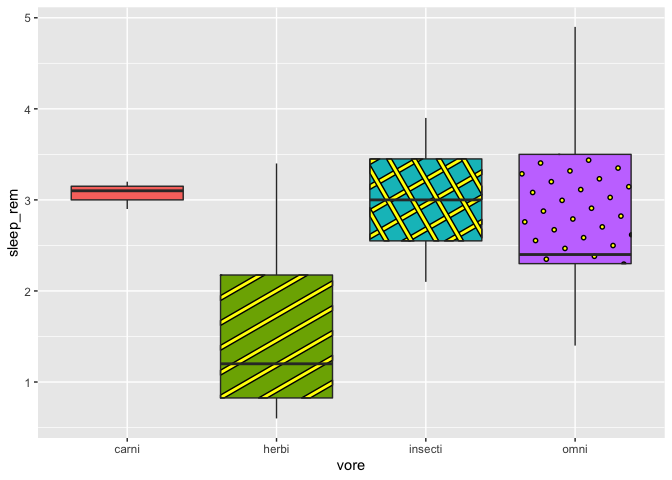
Attention, ici j’ai dû enlever la légende, car elle ne fonctionne que si l’on utilise pattern, comme l’argument fill ou l’argument color d’un geom régulier de ggplot2. En fait, ggpattern fonctionne presque qu’exactement comme ggplot2, à quelques exceptions près…comme le fait de pouvoir ajouter des images comme motif.
ggplot(msleep2, aes(x = vore, y = sleep_rem, fill = vore)) +
geom_boxplot_pattern(stat = "boxplot",
pattern = "image",
pattern_filename = "https://static.wikia.nocookie.net/orville/images/9/9c/BortusCropped.jpg/revision/latest?cb=20190106062857")
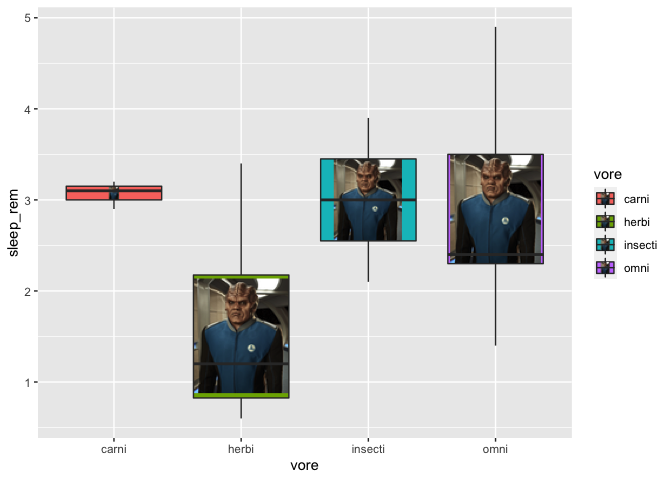
Ici, j’ai utilisé une image d’internet, mais je vous conseille de prendre des images enregistrées sur votre ordinateur, au cas où l’URL ne fonctionnerait plus. De plus, vous pourrez avoir une image différente pour chaque groupe de cette façon. À vous de vous amuser avec cette fonctionnalité!
Modifier TOUTES les étiquettes
Avec ggplot, toutes les étiquettes présentes dans un graphique peuvent être modifiées, même certaines que vous ne savez peut-être pas qu’elles existent. La clé est de savoir leur nom :
ggplot(msleep, aes(x = sleep_total, y = sleep_rem)) +
geom_point(aes(color = brainwt/bodywt)) +
scale_color_gradient2(
high = "#192168",
mid = "#ffffff",
low = "#af1e2d", # remarquez que l'on peut utiliser majuscules ou minuscules ici, sans problèmes
midpoint = 0.02
) +labs(
title = "Titre du grahpique",
subtitle = "Sous titre",
caption = "Source",
tag = "a)",
color = "Couleur",
x = "Axe des X",
y = "Axe des Y"
)
Warning: Removed 22 rows containing missing values (`geom_point()`).
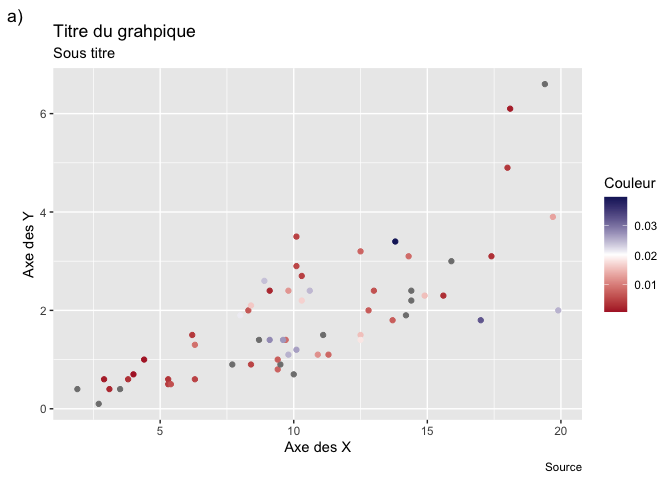
Remarquez que l’on utilisera rarement title, subtitle et caption, puisqu’il est plus commun d’entrer ces éléments dans l’éditeur de texte. Remarquez aussi que vous avez accès à tous les autres éléments de la légende de la même façon (shape, fill, etc.)
Les nuances de l’échelle log
Je vais maintenant discuter avec vous d’une opération somme toute banale, mais qui peut comporter plusieurs subtilités : mettre un axe en log.
ggplot(msleep, aes(x = bodywt, y = brainwt)) +
geom_point() +
scale_y_log10() +
scale_x_log10()
Warning: Removed 27 rows containing missing values (`geom_point()`).
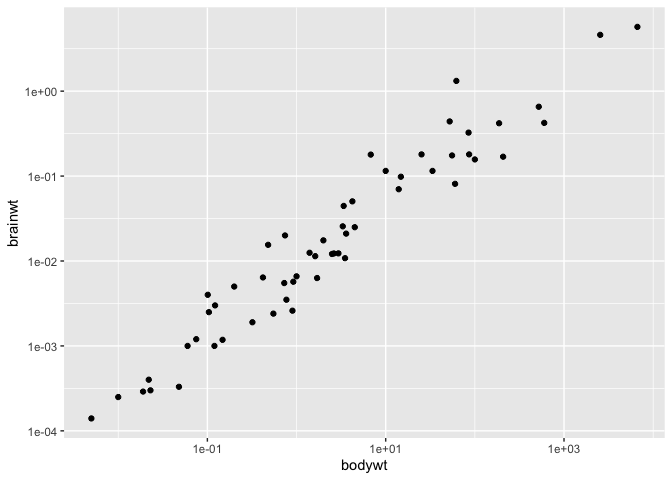
Ces fonctions sont un raccourci, plutôt que de transformer manuellement vos données comme ceci :
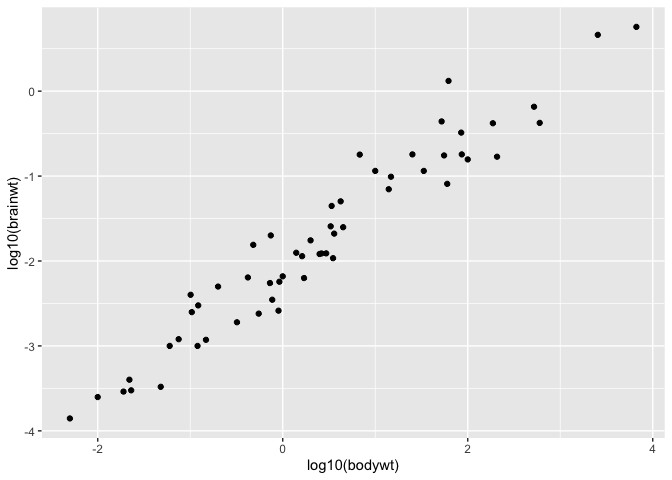
ggplot(msleep, aes(x = log10(bodywt), y = log10(brainwt))) +
geom_point()
Warning: Removed 27 rows containing missing values (`geom_point()`).
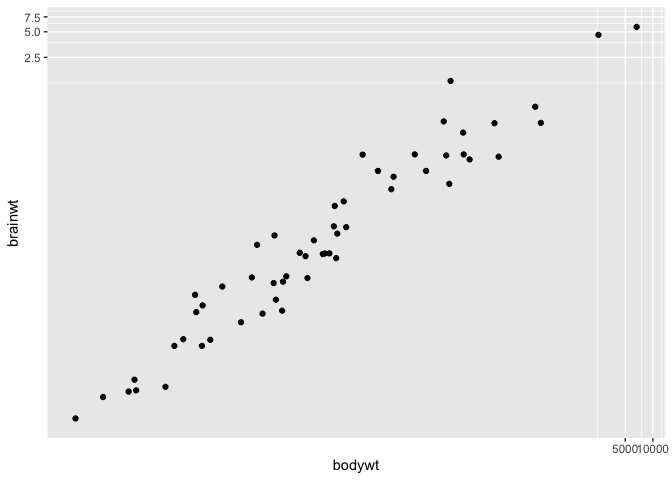
Il pourrait cependant arriver que plutôt que transformer les valeurs, on ait besoin de seulement transformer l’axe, sans toucher aux valeurs comme tel. Il faut à ce moment utiliser coord_trans :
ggplot(msleep, aes(x = bodywt, y = brainwt)) +
geom_point() +
coord_trans(x = "log10", y = "log10")
Warning: Removed 27 rows containing missing values (`geom_point()`).
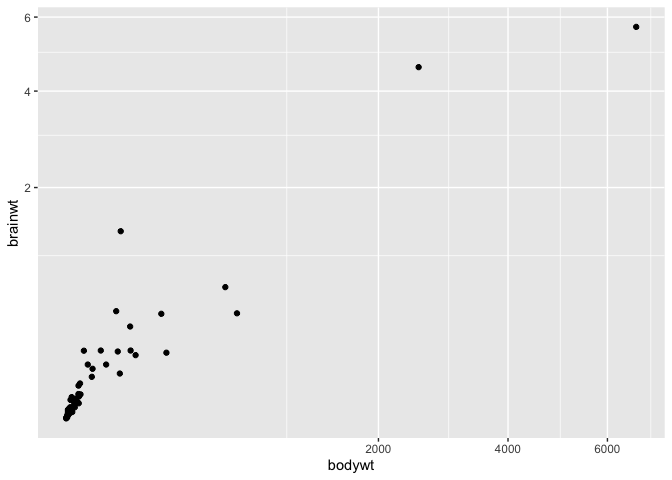
Vous pouvez entrer dans la transformation n’importe quelle fonction, par exemple la racine carrée :
ggplot(msleep, aes(x = bodywt, y = brainwt)) +
geom_point() +
coord_trans(x = "sqrt", y = "sqrt")
Warning: Removed 27 rows containing missing values (`geom_point()`).
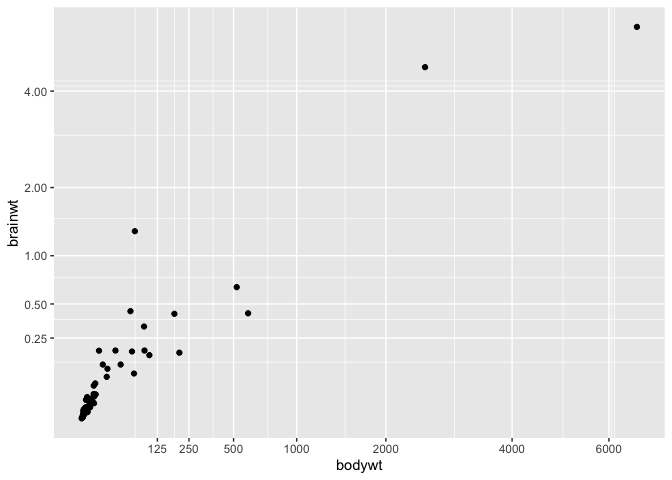
Modifier les marques sur un axe
J’en profite ici pour vous montrer comment modifier manuellement les valeurs utilisées comme tick marks sur les axes.
ggplot(msleep, aes(x = bodywt, y = brainwt)) +
geom_point() +
coord_trans(x = "sqrt", y = "sqrt") +
scale_x_continuous(breaks = c(125,250,500,1000,2000,4000,6000)) +
scale_y_continuous(breaks = c(0.25,0.5,1,2,4,8))
Warning: Removed 27 rows containing missing values (`geom_point()`).
Pour la publication
Combiner plusieurs graphiques
Une des questions de ggplot2 que je dois me faire poser le plus souvent est : comment combiner plusieurs graphiques dans une même image. La difficulté de cette question tient, entre autres, au fait que ce n’est pas une fonction directe de ggplot2. Elle provient de librairies externes, donc il existe de multiples façons de faire. Et ce n’est pas toutes les façons qui permettent d’utiliser correctement ggsave à la fin de tout pour produire un fichier haute-résolution.
La méthode que je vous propose ici est une nouvelle (2023) et robuste à la sauvegarde avec ggsave. Vous aurez besoin pour y arriver de la librairie patchwork.
On prépare d’abord nos graphiques individuellement et on les place dans un objet:
g1 <- ggplot(msleep, aes(x = log(bodywt), y = awake)) + geom_point() +labs(tag = "a)")
g1
g2 <- ggplot(msleep, aes(x = awake)) + geom_histogram() +labs(tag = "b)")
g2
`stat_bin()` using `bins = 30`. Pick better value with `binwidth`.
g3 <- ggplot(msleep, aes(x = log(bodywt))) + geom_histogram() +labs(tag = "c)")
g3
`stat_bin()` using `bins = 30`. Pick better value with `binwidth`.
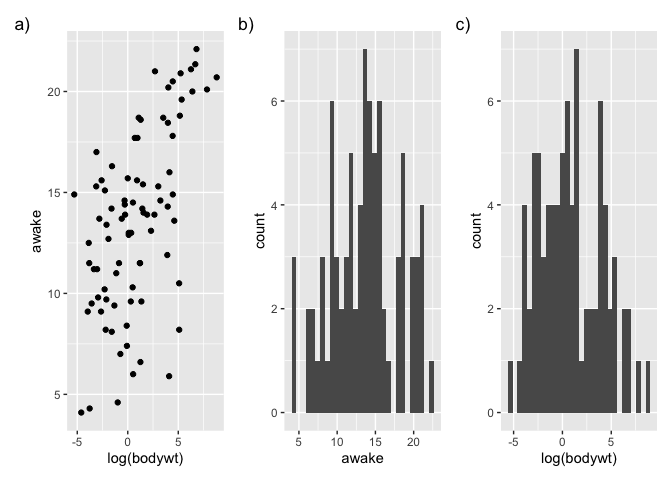
Puis, l’opérateur + nous permet de les combiner :
library(patchwork)
Warning: package 'patchwork' was built under R version 4.3.1
p <- g1 + g2 + g3
p
`stat_bin()` using `bins = 30`. Pick better value with `binwidth`.
`stat_bin()` using `bins = 30`. Pick better value with `binwidth`.
ggsave("test.jpg", p, width = 9, height = 4, dpi = 300) # Pour sauvegarder le graphique
`stat_bin()` using `bins = 30`. Pick better value with `binwidth`.
`stat_bin()` using `bins = 30`. Pick better value with `binwidth`.
Notez que la librarie patchwork a ajouté une nouvelle fonctionnalité à l’opérateur +. Nous n’avons donc pas eu à faire appel à une nouvelle fonction.
On peut également se préparer des layouts plus complexes :
p <- (g1 | g2) / g3
p
`stat_bin()` using `bins = 30`. Pick better value with `binwidth`.
`stat_bin()` using `bins = 30`. Pick better value with `binwidth`.
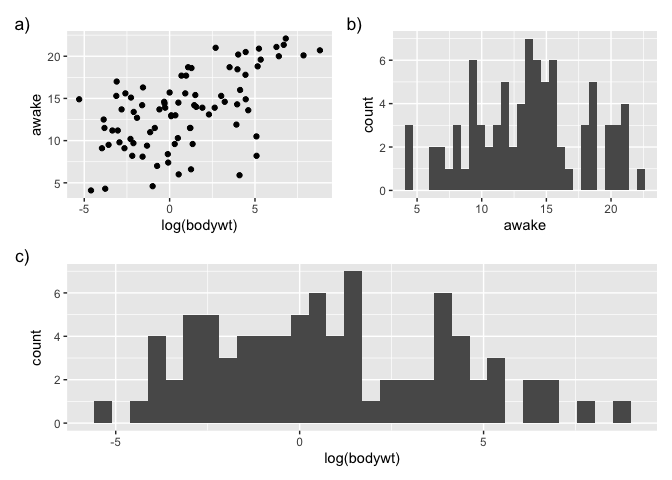
ggsave("test2.jpg", p, width = 4, height = 4, dpi = 300)
`stat_bin()` using `bins = 30`. Pick better value with `binwidth`.
`stat_bin()` using `bins = 30`. Pick better value with `binwidth`.
En explorant la librarie, il y a aussi moyen de spécifier la hauteur des graphiques individuellement, d’ajouter un titre ou encore une légende commune.
Changer l’ordre de variables qualitatives
Une chose qui vous arrivera de temps à autre au moment de finaliser vos graphiques avant publication sera de vouloir modifier l’ordre des éléments associés à une variables qualitative, par exemple dans l’axe des X d’un boxplot.
Prenons ce petit graphique comme exemple :
ggplot(msleep, aes(x = vore, y = sleep_rem)) +
geom_boxplot()
Warning: Removed 22 rows containing non-finite values (`stat_boxplot()`).
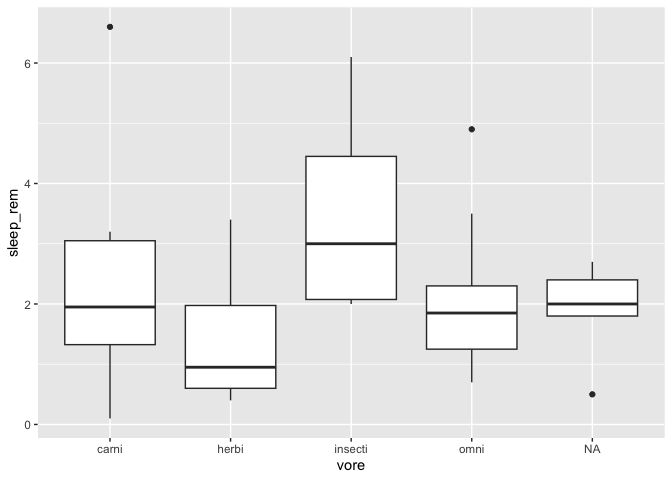
Une des choses que l’on pourrait vouloir changer dans ce graphique est de trier les types d’alimentation du plus petit au plus grand. En particulier si ce graphique est associé à un test statistique de type ANOVA.
La façon la plus simple de faire est de modifier l’ordre de notre variable catégorique dans le tableau de données avant de l’envoyer au grahpique. Il existe dans le tidyverse une librairie faite exprès pour manipuler les variables catégoriques, qui se nomme forcats.
Voyons comment changer l’ordre de vore pour qu’il soit basé sur la moyenne du temps de sommeil paradoxal :
library(forcats)
library(tidyr)
msleep %>%
drop_na() %>%
mutate(vore = fct_reorder(vore,sleep_rem)) %>%
ggplot(aes(x = vore, y = sleep_rem)) +
geom_boxplot()
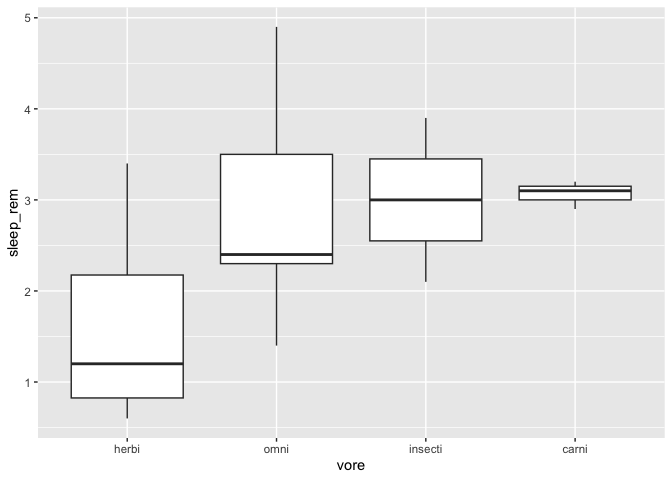
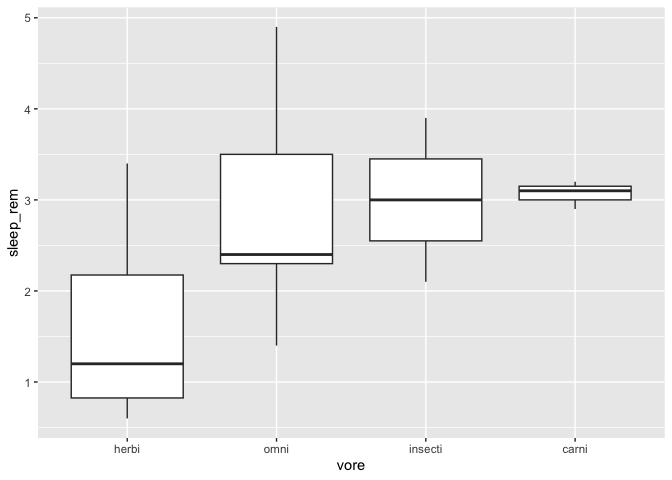
Si on me mentionne rien, le tri se fait par la médiane des valeurs. On peut aussi spécifier une autre fonction, par exemple trier par la moyenne :
msleep %>%
drop_na() %>%
mutate(vore = fct_reorder(vore,sleep_rem,mean)) %>%
ggplot(aes(x = vore, y = sleep_rem)) +
geom_boxplot()
On peut aussi choisir manuellement l’ordre des bandes manuellement, avec la fonction fct_relevel.
msleep %>%
drop_na() %>%
mutate(vore = fct_relevel(vore,"omni","insecti")) %>%
ggplot(aes(x = vore, y = sleep_rem)) +
geom_boxplot()
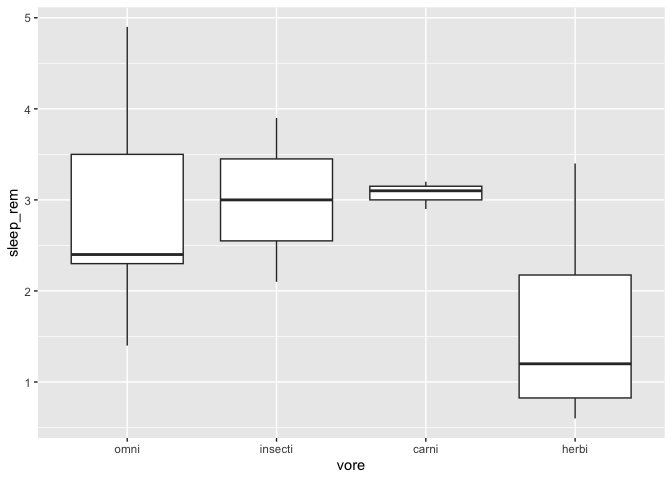
Les niveaux non-mentionnés sont ajoutés à la fin de la liste, dans le même ordre qu’originalement.
Ajouter de petits icônes ou images
Il est aussi possible d’ajouter de petits icônes (ou des grosses images!) à vos graphiques, en téléchargeant la librairie ggimage. Cette dernière ajoute un geom, nommé geom_image qui permet d’insérer des images.
library(ggimage)
msleep %>%
drop_na() %>%
mutate(vore = fct_relevel(vore,"omni","insecti")) %>%
ggplot(aes(x = vore, y = sleep_rem)) +
geom_boxplot() +
geom_image(size = 0.08, aes(x = "herbi", y = 5.2, image = "https://static.thenounproject.com/png/2545-200.png")) +
geom_image(size = 0.08,aes(x = "carni", y = 5.2, image = "https://static.thenounproject.com/png/18664-200.png")) +
geom_image(size = 0.08,aes(x = "omni", y = 5.2, image = "https://static.thenounproject.com/png/5271-200.png")) +
geom_image(size = 0.08,aes(x = "insecti", y = 5.2, image = "https://static.thenounproject.com/png/2546-200.png"))
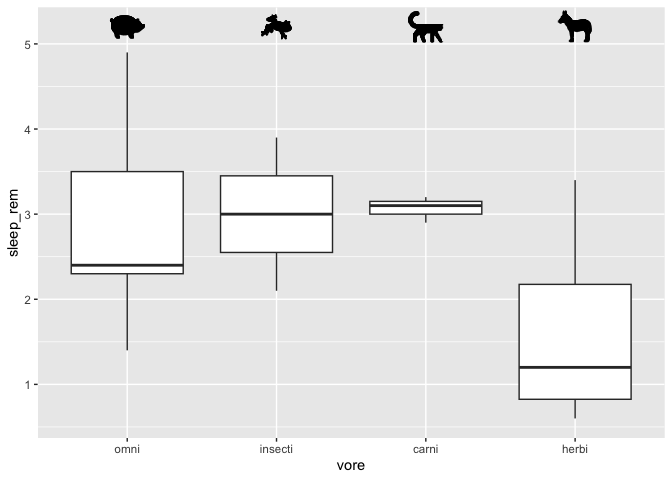
Remarquez qu’il serait préférable de placer les images sur votre ordinateur et de fournir à R un chemin du genre c:\Windows\etc. car si l’image disparaît du site, votre graphique ne fonctionnera plus.
Remarquez aussi que, si vous avez plus de 2-3 images, il pourrait être préférable de stocker cette information dans un tableau de données plutôt que de tout faire à la main dans la graphique comme j’ai fait.
Enfin, ces images auraient été de bonnes candidates à la fonction annotate, mais au moment de terminer la formation, ça ne fonctionnait toujours pas. Peut-être que annotate ne fonctionne qu’avec les geom par défaut de ggplot2? À voir…
Autres / Misc.
Tracer n’importe quelle forme
Si vous avez besoin de dessiner quelque chose en particulier qui n’est pas déjà défini par un geom, sachez que vous pouvez préparer une série de coordonnées, et les faire connecter par un geom_path, par exemple comme ceci :
data.frame(
x = c(1,2,3,1),
y = c(1,2,1,1)
) %>%
ggplot(aes(x = x, y = y)) +
geom_path()
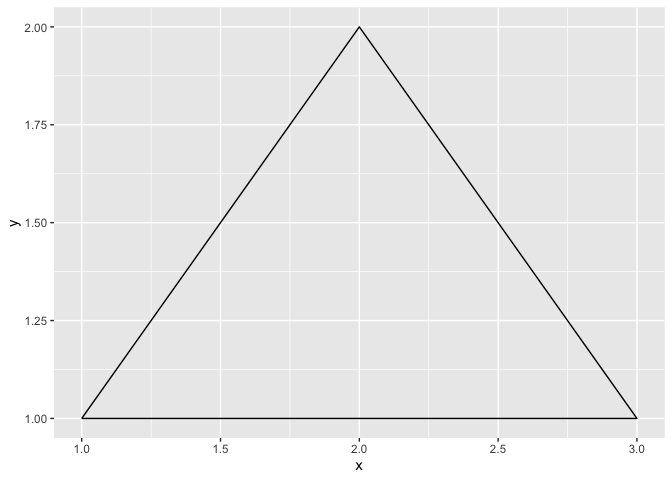
Ça peut demander un peu de réflexion, mais vous pouvez vraiment faire CE QUE VOUS VOULEZ!
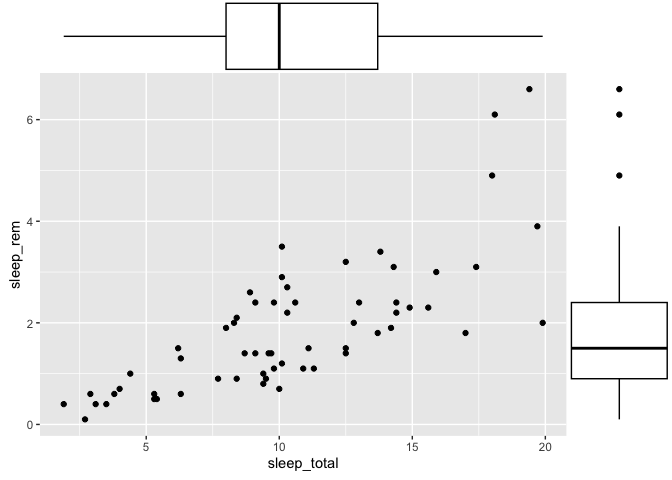
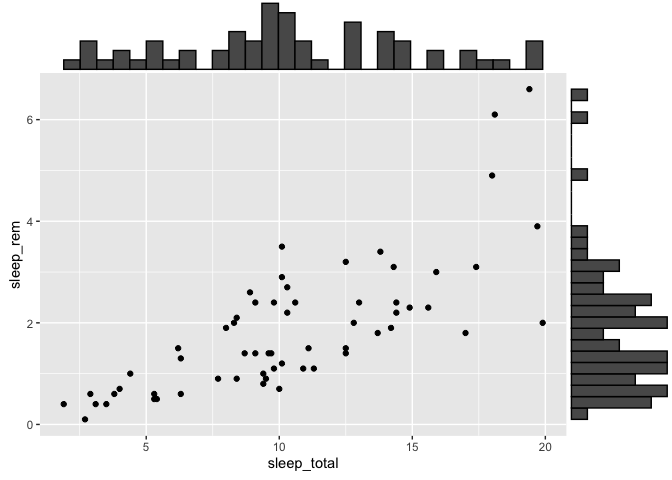
Distributions marginales
Si jamais vous avez besoin d’ajouter des distributions marginales à un graphique avec ggplot2, sachez qu’il existe une fonction exprès pour cela dans la librairie ggExtra, qui se nomme ggMarginal. Elle s’utilise comme ceci :
library(ggExtra)
p <- ggplot(msleep, aes(x = sleep_total, y = sleep_rem)) +
geom_point()
ggMarginal(p, type = "histogram")
Warning: Removed 22 rows containing missing values (`geom_point()`).
ggMarginal(p, type = "boxplot")
Warning: Removed 22 rows containing missing values (`geom_point()`).